[Python] LLE 를 통한 차원 축소 (dimensionality reduction by Locally Linear Embedding, LLE)
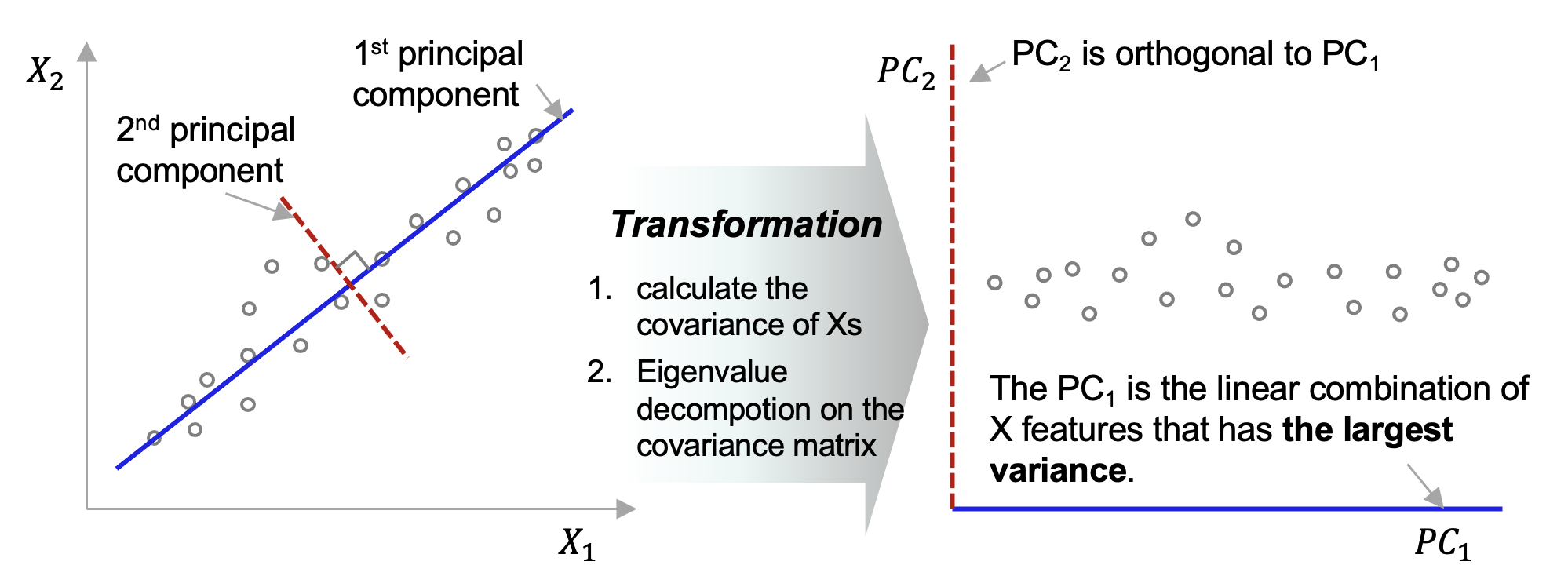
Python 분석과 프로그래밍/Python 기계학습 2022. 9. 18. 22:46지난번 포스팅에서는 차원 축소란 무엇이고 왜 하는지(https://rfriend.tistory.com/736)와 투영(projection) 방법을 사용하는 주성분 분석(PCA)을 통한 차원 축소 (https://rfriend.tistory.com/751) 에 대해서 소개하였습니다.
이번 포스팅에서는 manifold learning approche 에 해당하는 비지도학습, 비선형 차원축소 방법으로서 LLE (Locally Linear Embedding) 알고리즘을 설명하겠습니다.
(1) 매니폴드 학습 (Manifold Learning)
(2) Locally Linear Embedding (LLE) 알고리즘
(3) sklearn 을 사용한 LLE 실습
(1) 매니폴드 학습 (Maniflod Learning)
먼저 매니폴드 학습(Manifold Learning)에 대해서 간략하게 살펴보고 넘어가겠습니다.
매니폴드란 각 점 근처의 유클리드 공간과 국부적으로 유사한 위상 공간을 말합니다. (A manifold is a topological space that locally resembles Eucludian space near each point.) [1] d-차원의 매니폴드는 n-차원 공간 (이때 d < n) 의 부분으로서 d-차원의 초평면(d-dimensional hyperplane)을 국부적으로 닮았습니다. 아래의 3차원 공간의 Swiss Roll 에서 데이터의 구성 특성과 속성을 잘 간직한 2차원의 평면(plane)을 생각해볼 수 있는데요, Swiss Roll 을 마치 돌돌 말린 종이(매니폴드)를 펼쳐서 2차원 공간으로 표현해본 것이 오른쪽 그림입니다.
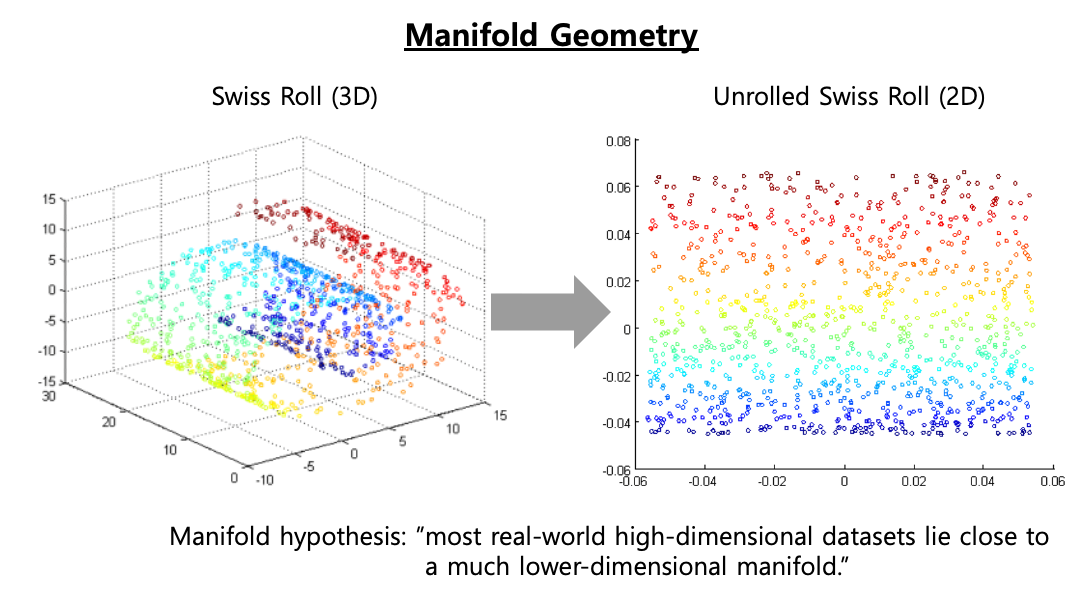
매니폴드 학습(Manifold Learning)이란 데이터에 존재하는 매니폴드를 모델링하여 차원을 축소하는 기법을 말합니다. 매니폴드 학습은 대부분 실세계의 고차원 데이터가 훨씬 저차원의 매니폴드에 가깝게 놓여있다는 매니폴드 가정(Manifold Assumption) 혹은 매니폴드 가설(Maniflod Hypothesis)에 기반하고 있습니다. [2]
위의 비선형인 3차원 Swiss Roll 데이터를 선형 투영 기반의 주성분 분석(PCA, Principal Component Analysis)이나 다차원척도법(MDS, Multi-Dimensional Scaling) 로 2차원으로 차원을 축소하려고 하면 데이터 내 존재하는 매너폴드를 학습하지 못하고 데이터가 뭉개지고 겹쳐지게 됩니다.
매니폴드 가설은 분류나 회귀모형과 같은 지도학습을 할 때 고차원의 데이터를 저차원의 매니폴드 공간으로 재표현했을 때 더 간단하고 성과가 좋을 것이라는 묵시적인 또 다른 가설과 종종 같이 가기도 합니다. 하지만 이는 항상 그런 것은 아니며, 데이터셋이 어떻게 구성되어 있느냐에 전적으로 의존합니다.
(2) Locally Linear Embedding (LLE) 알고리즘
LLE 알고리즘은 "Nonlinear Dimensionality Reduction by Locally Linear Embedding (2000)" [3] 논문에서 주요 내용을 간추려서 소개하겠습니다.
LLE 알고리즘은 주성분분석이나(PCA) 다차원척도법(MDS)와는 달리 광범위하게 흩어져있는 데이터 점들 간의 쌍을 이룬 거리를 추정할 필요가 없습니다. LLE는 국소적인 선형 적합으로 부터 전역적인 비선형 구조를 복원합니다 (LLE revocers global nonlinear structure from locally linear fits.).
논문에서 소개한 LLE 3단계 절차(steps of locally linear embedding)는 꽤 간단합니다.
(1단계) 각 데이터 점의 이웃을 선택 (Select neighbors)
(2단계) 이웃으로부터 선형적으로 가장 잘 재구성하는 가중치를 계산 (Reconstruct with linear weights)
(3단계) 가중치를 사용해 저차원의 임베딩 좌표로 매핑 (Map to embedded coordinates)
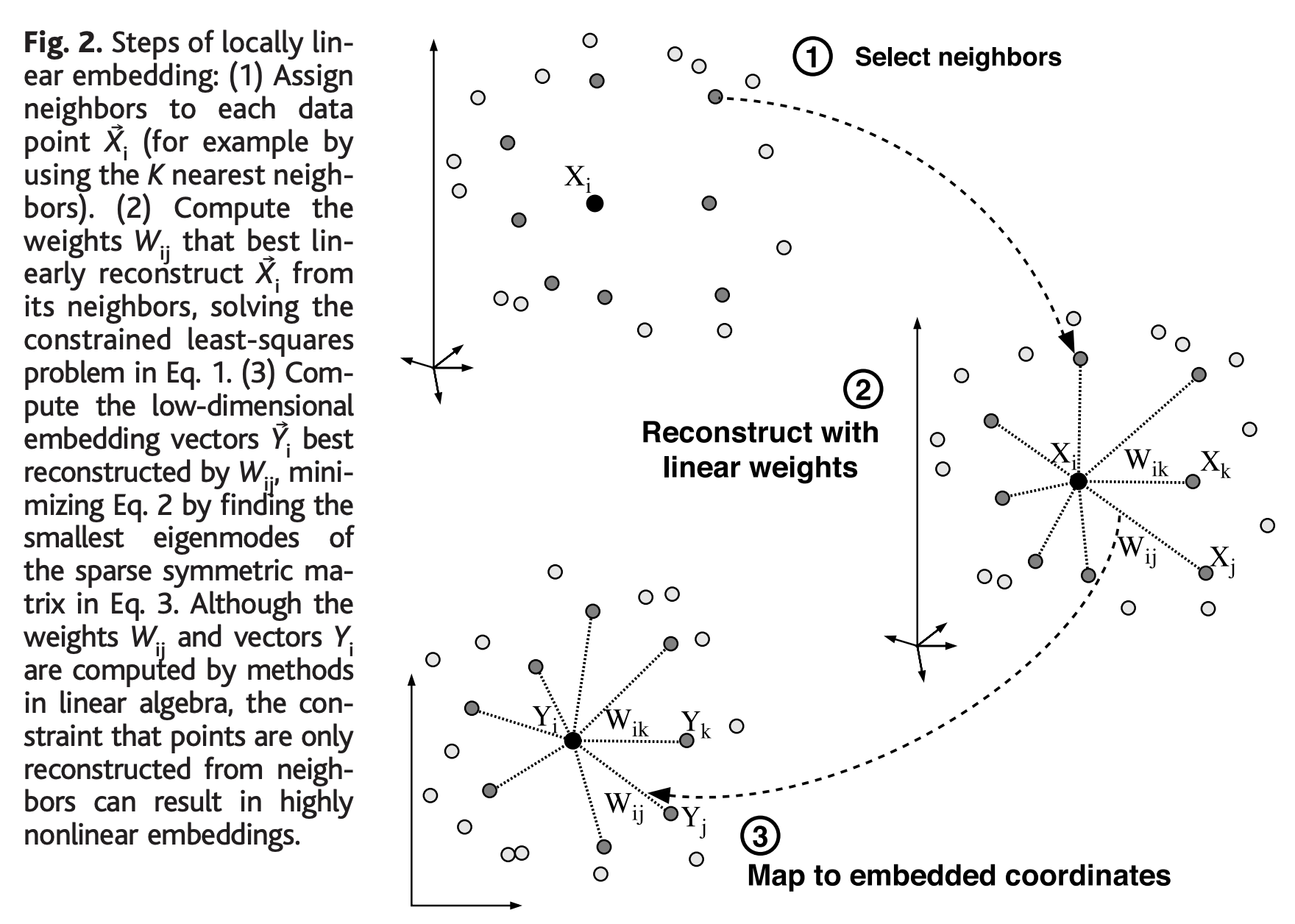
1단계에서 각 데이터 점별로 이웃을 할당할 때는 데이터 점들 간의 거리를 계산하는데요, 가령 K 최근접이웃(K nearest neighbors) 기법을 사용할 수 있습니다.
2단계에서 각 데이터 점들의 이웃들로부터 각 점을 가장 잘 재구성하는 선형 회귀계수(linear coefficients, linear weights)를 계산해서 국소적인 기하 특성을 간직한 매너폴드를 학습합니다. 아래는 재구성 에러를 측정하는 비용함수인데요, 원래의 데이터점과 이웃들로 부터 계산한 선형 모형으로 재구성한 값과의 거리를 제곱하여 모두 더한 값입니다.
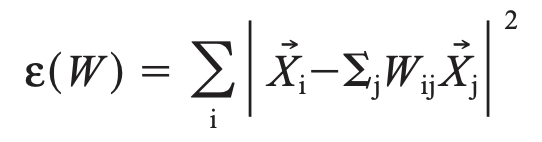
위의 비용함수를 최소로 하는 가중치 벡터 Wij 를 계산하게 되는데요, 이때 2가지 제약조건(constraints)이 있습니다.
- (a) 각 데이터 점은 단지 그들의 이웃들로 부터만 재구성됩니다.
(만약 Xj 가 Xi의 이웃에 속하지 않는 데이터점이면 가중치 Wij = 0 이 됩니다.)
- (b) 가중치 행렬 행의 합은 1이 됩니다. (sum(Wij) = 1)
위의 2가지 제약조건을 만족하면서 비용함수를 최소로 하는 가중치 Wij 를 구하면 특정 데이터 점에 대해 회전(rotations), 스케일 조정(recalings), 그리고 해당 데이터 점과 인접한 데이터 점의 변환(translations) 에 있어 대칭(symmetry)을 따릅니다. 이 대칭에 의해서 (특정 참조 틀에 의존하는 방법과는 달리) LLE의 재구성 가중치는 각 이웃 데이터들에 내재하는 고유한 기하학적 특성(저차원의 매너폴드)을 모델링할 수 있게됩니다.
3단계에서는 고차원(D) 벡터의 각 데이터 점 Xi 를 위의 2단계에서 계산한 가중치를 사용하여 매니폴드 위에 전역적인 내부 좌표를 표현하는 저차원(d) 벡터 Yi 로 매핑합니다. 이것은 아래의 임베팅 비용 함수를 최소로 하는 저차원 좌표 Yi (d-mimensional coordinates) 를 선택하는 것으로 수행됩니다.

임베팅 비용 함수는 이전의 선형 가중치 비용함수와 마찬가지로 국소 선형 재구성 오차를 기반으로 합니다. 하지만 여기서 우리는 Yi 좌표를 최적화하는 동안 선형 가중치 Wij 를 고정합니다. 임베팅 비용 함수는 희소 N x N 고유값 문제(a sparse N X N eignevalue problem)를 풀어서 최소화할 수 있습니다. 이 선형대수 문제를 풀면 하단의 d차원의 0이 아닌 고객벡터는 원점을 중심으로 정렬된 직교 좌표 집합을 제공합니다.
LLE 알고리즘은 희소 행렬 알고리듬(sparse matrix algorithms)을 활용하기 위해 구현될 때 비선형 차원축소 기법 중 하나인 Isomap 보다 더 빠른 최적화와 많은 문제에 대해 더 나은 결과를 얻을 수 있는 몇 가지 장점이 있습니다. [4]
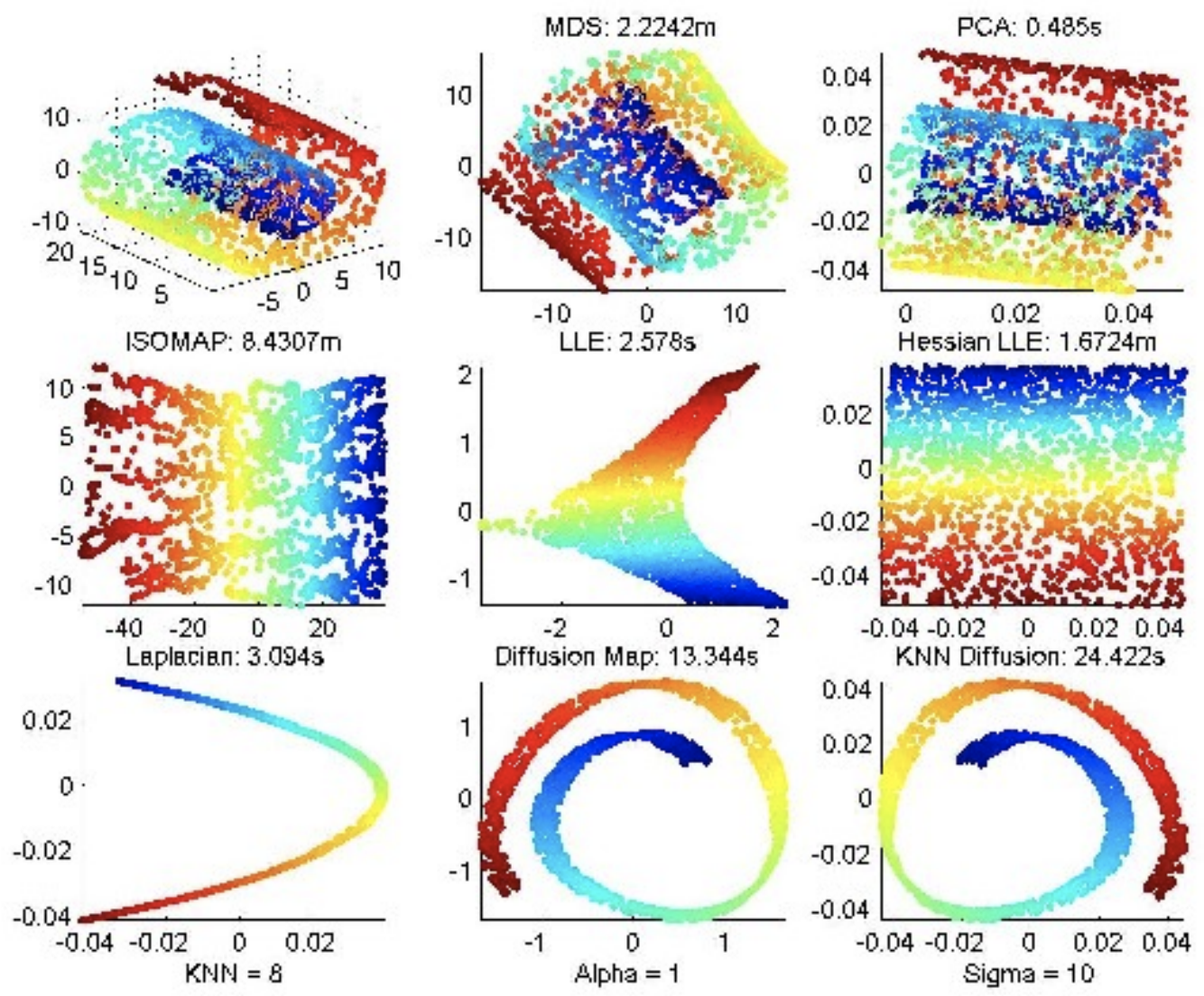
아래에 3차원 Swiss Roll 데이터를 여러가지 비선형 차원 축소 기법을 사용해서 적용한 결과인데요 [5], LLE 는 수행 시간이 짧은 장점이 있지만 매니폴드가 약간 찌그러져 있는 한계가 있는 반면에, Isomap과 Hessian LLE 는 국지적인 데이터 형상과 관계를 잘 재표현한 저차원 매니폴드를 잘 잡아내지만 수행 시간이 LLE 대비 굉장히 오래걸리는 단점이 있습니다.
(3) sklearn 을 사용한 LLE 실습
먼저 sklearn 모듈의 make_swiss_roll 메소드를 사용해서 데이터 점 1,000개를 가지는 3차원의 Swiss Roll 샘플 데이터셋을 만들어보겠습니다. X 는 데이터 점이고, t는 매니폴드 내 점의 주 차원에 따른 샘플의 단변량 위치입니다.
## Swiss Roll sample dataset
## ref: https://scikit-learn.org/stable/modules/generated/sklearn.datasets.make_swiss_roll.html
from sklearn.datasets import make_swiss_roll
X, t = make_swiss_roll(n_samples=1000, noise=0.1, random_state=1004)
## X: The points.
X[:5]
# array([[ 1.8743272 , 18.2196214 , -4.75535504],
# [12.43382272, 13.9545544 , 2.91609936],
# [ 8.02375359, 14.23271056, -8.67338106],
# [12.23095692, 2.37167446, 3.64973091],
# [-8.44058318, 15.47560926, -5.46533069]])
## t: The univariate position of the sample according to the main dimension of the points in the manifold.
t[:10]
# array([ 5.07949952, 12.78467229, 11.74798863, 12.85471755, 9.99011767,
# 5.47092408, 6.89550966, 6.99567358, 10.51333994, 10.43425738])
다음으로 sklearn 모듈에 있는 LocallyLinearEmbedding 메소드를 사용해서 위에서 생성한 Swiss Roll 데이터에 대해 LLE 알고리즘을 적용하여 2차원 데이터로 변환을 해보겠습니다.
## Manifold Learning
from sklearn.manifold import LocallyLinearEmbedding
lle = LocallyLinearEmbedding(n_components=2, n_neighbors=10)
X_reduced_lle = lle.fit_transform(X)
X_reduced_lle[:10]
# array([[-0.0442308 , 0.0634603 ],
# [ 0.04241534, 0.01060574],
# [ 0.02712308, 0.01121903],
# [ 0.04396569, -0.01883799],
# [ 0.00275144, 0.01550906],
# [-0.04178513, 0.05415933],
# [-0.03073913, 0.023496 ],
# [-0.02880368, -0.0230327 ],
# [ 0.0109238 , -0.02566617],
# [ 0.00979253, -0.02309815]])
마지막으로 matplotlib 모듈을 사용해서 2차원 평면에 위에서 LLE 로 매핑한 데이터를 시각화해보겠습니다. 색깔을 t 로 구분하였습니다.
## 2D Scatter Plot
import matplotlib.pyplot as plt
plt.rcParams['figure.figsize'] = (8, 8)
plt.scatter(X_reduced_lle[:, 0], X_reduced_lle[:, 1], c=t, cmap=plt.cm.hot)
plt.title("Unrolled Swiss Roll by LLE", fontsize=20)
plt.xlabel("$y_1$", fontsize=14)
plt.ylabel("$y_2$", fontsize=14)
plt.axis([-0.065, 0.055, -0.1, 0.12])
plt.grid(True)
plt.show()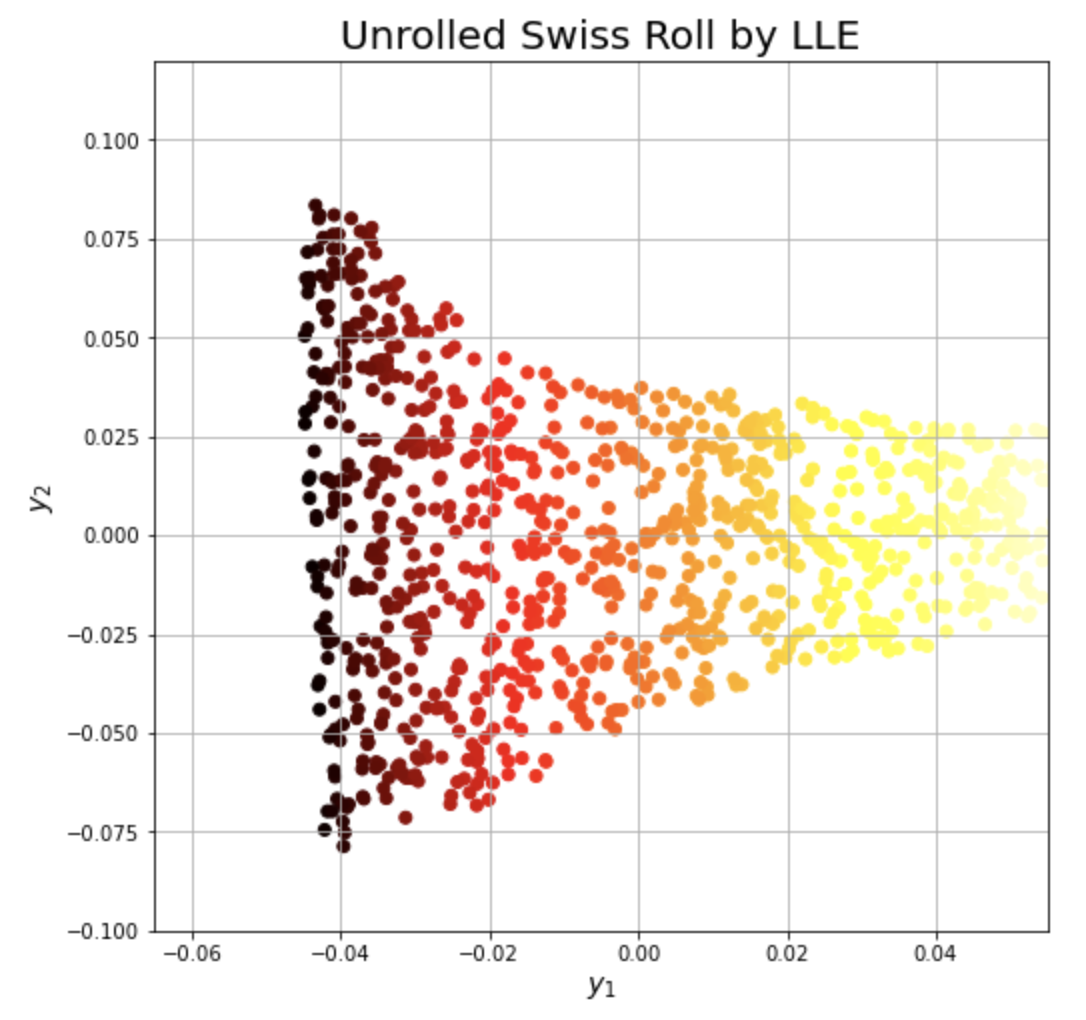
[Reference]
[1] Wikipedia, "Manifold", https://en.wikipedia.org/wiki/Manifold
[2] Aurelien Geron, "Hands-On Machine Learning with Scikit-Learn & Tensorflow"
[3] Sam T. Roweis, Lawrence K. Saul, "Nonlinear Dimensionality Reduction by Locally Linear Embedding"
[4] Wikipedia, "Nonlinear Dimensionality Reduction", https://en.wikipedia.org/wiki/Nonlinear_dimensionality_reduction
[5] Nik Melchior, "Manifold Learning, Isomap and LLE": https://www.cs.cmu.edu/~efros/courses/AP06/presentations/melchior_isomap_demo.pdf
이번 포스팅이 많은 도움이 되었기를 바랍니다.
행복한 데이터 과학자 되세요! :-)