[Greenplum] PL/Python을 이용해서 병렬로 시계열 데이터 결측값 보간하기 (interpolation in parallel using PL/Python on Greenplum)
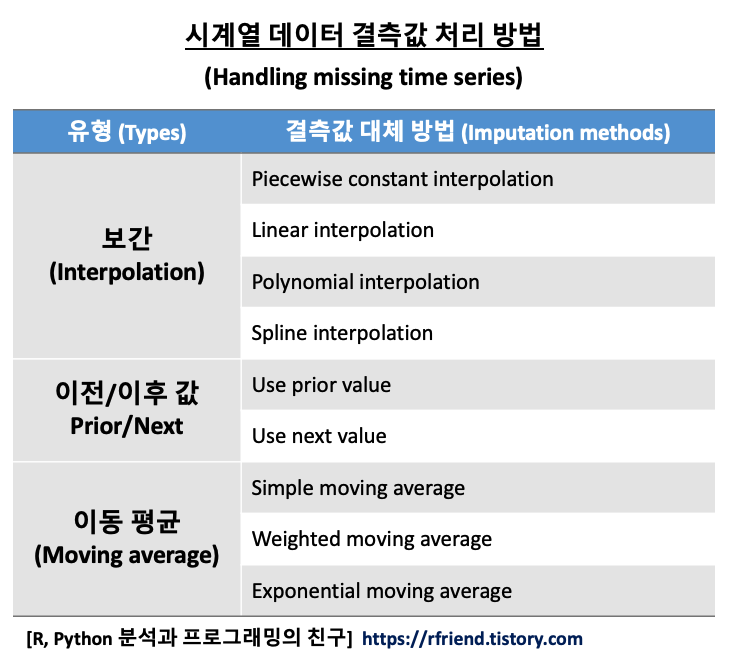
Greenplum and PostgreSQL Database 2023. 1. 23. 14:02시계열 데이터를 분석할 때 제일 처음 확인하고 처리하는 일이 결측값(missing values) 입니다. 이번 포스팅에서는 시계열 데이터의 결측값을 선형 보간(linear interpolation)하는 2가지 방법을 소개하겠습니다.
(1) Python 으로 결측값을 포함하는 예제 시계열 데이터 생성하기
(2) Python 의 for loop 순환문으로 시계열 데이터 결측값을 보간하기
(interpolation sequentially using Python for loop statement)
(3) Greenplum에서 PL/Python으로 분산병렬처리하여 시계열 데이터 결측값을 보간하기
(interpolation in parallel using PL/Python on Greenplum)
(1) Python 으로 결측값을 포함하는 예제 시계열 데이터 생성하기
샘플 시계열 데이터를 저장할 폴더를 base_dir 에 지정해주었습니다.
분석 단위로 사용할 Lot, Cell, Parameter, TimeStamp 의 개수를 지정해 줍니다. 아래 예에서는 각 Lot, Cell, Parameter 별로 100개의 TimeStamp 별로 측정값을 수집하고, 각 분석 단위별로 10개의 결측값을 포함하도록 설정했습니다.
np.random.normal(10, 30, ts_num)
: 측정값은 정규분포 X~N(10, 3) 의 분포로 부터 ts_num 인 100개를 난수 생성하여 만들었습니다.
nan_mask = np.random.choice(np.arange(ts_num), missing_num)
ts_df_tmp.loc[nan_mask, 'measure_val'] = np.nan
: 각 분석 단위의 100 개의 측정치 중에서 무작위로 missing_num = 10 개를 뽑아서 np.nan 으로 교체하여 결측값으로 변경하였습니다.
하나의 Lot에 Cell 100개, 각 Cell별 Parameter 10개, 각 Parameter 별 TimeStamp의 측정치 100개, 이중 결측치 10개를 포함한 시계열 데이터를 Lot 별로 묶어서(concat) DataFrame을 만들고, 이를 CSV 파일로 내보냅니다.
#%% setting the directories and conditions
base_dir = '/Users/Documents/ts_data/'
## setting the number of IDs' conditions
lot_num = 1000
cell_num = 100
param_num = 10
missing_num = 10
ts_num = 100 # number of TimeStamps
#%% [Step 1] generating the sample dataset
import numpy as np
import pandas as pd
import os
from itertools import chain, repeat
## defining the UDF
def ts_random_generator(lot_id, cell_num, param_num, ts_num, missing_num, base_dir):
# blank DataFrame for saving the sample datasets later
ts_df = pd.DataFrame()
for cell_id in np.arange(cell_num):
for param_id in np.arange(param_num):
# making a DataFrame with colums of lot_id, cell_cd, param_id, ts_id, and measure_val
ts_df_tmp = pd.DataFrame({
'lot_id': list(chain.from_iterable(repeat([lot_id + 1], ts_num))),
'cell_id': list(chain.from_iterable(repeat([cell_id + 1], ts_num))),
'param_id': list(chain.from_iterable(repeat([param_id + 1], ts_num))),
'timestamp_id': (np.arange(ts_num) + 1),
'measure_val': np.random.normal(10, 3, ts_num)# X~N(mean, stddev, size)
})
# inserting the missing values randomly
nan_mask = np.random.choice(np.arange(ts_num), missing_num)
ts_df_tmp.loc[nan_mask, 'measure_val'] = np.nan
# concatenate the generated random dataset(ts_df_tmp) to the lot based DataFrame(ts_df)
ts_df = pd.concat([ts_df, ts_df_tmp], axis=0)
# exporting the DataFrame to local csv file
base_dir = base_dir
file_nm = 'lot_' + \
str(lot_id+1).zfill(4) + \
'.csv'
ts_df.to_csv(os.path.join(base_dir, file_nm), index=False)
#ts_df.to_csv('/Users/lhongdon/Documents/SK_ON_PoC/ts_data/lot_0001.csv')
print(file_nm, "is successfully generated.")
#%% Executing the ts_random_generator UDF above
## running the UDF above using for loop statement
for lot_id in np.arange(lot_num):
ts_random_generator(
lot_id,
cell_num,
param_num,
ts_num,
missing_num,
base_dir
)
위의 코드를 실행하면 for loop 순환문이 lot_num 수만큼 돌면서 ts_random_generator() 사용자 정의함수를 실행시키면서 결측값을 포함한 시계열 데이터 샘플 CSV 파일을 생성하여 지정된 base_dir 폴더에 저장을 합니다.
(아래 화면 캡쳐 참조)
아래의 화면캡쳐는 결측값을 포함하는 시계열 데이터 샘플 중에서 LOT_0001 번의 예시입니다.
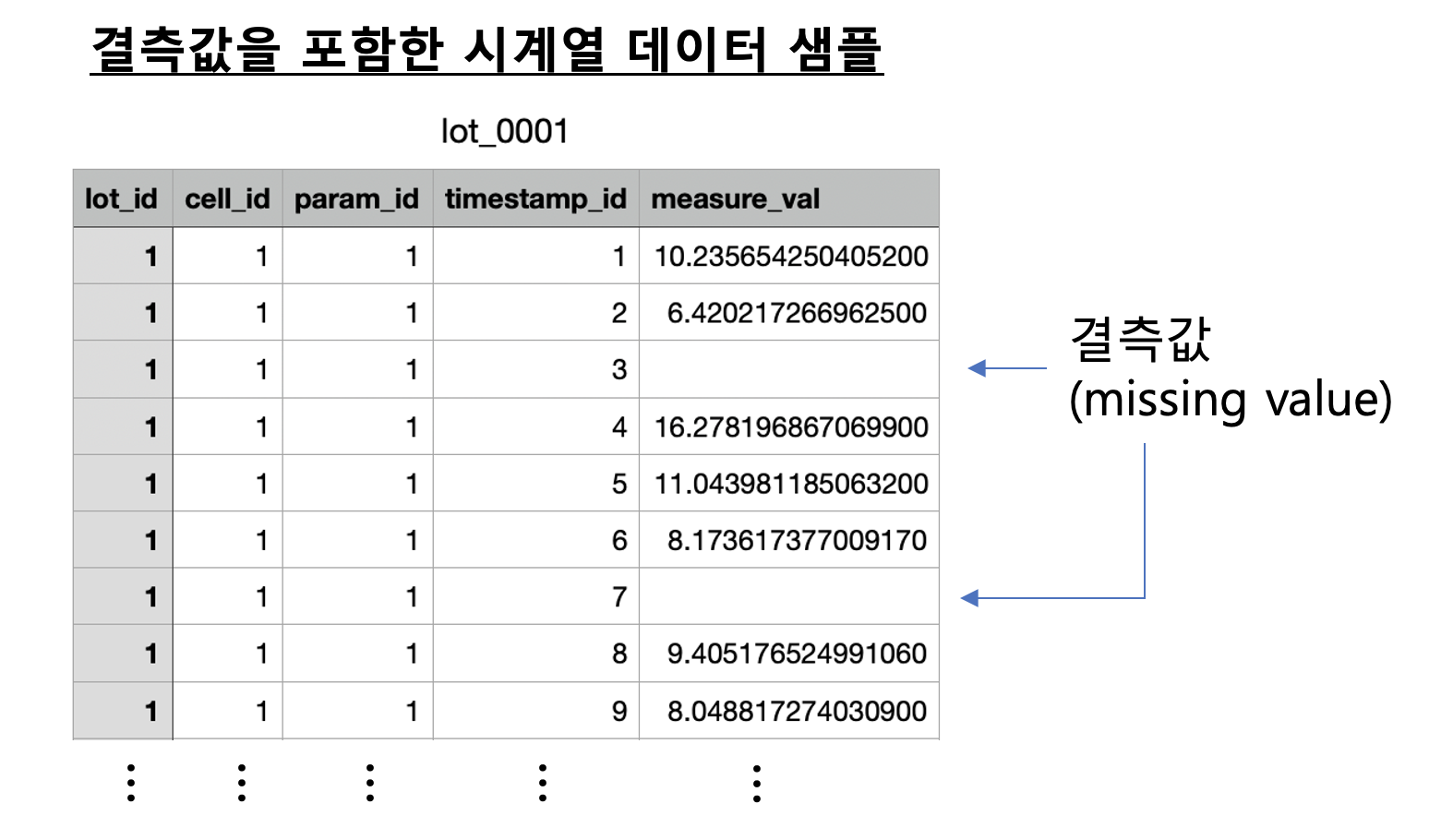
(2) Python 의 for loop 순환문으로 시계열 데이터 결측값을 보간하기
(interpolation sequentially using Python for loop statement)
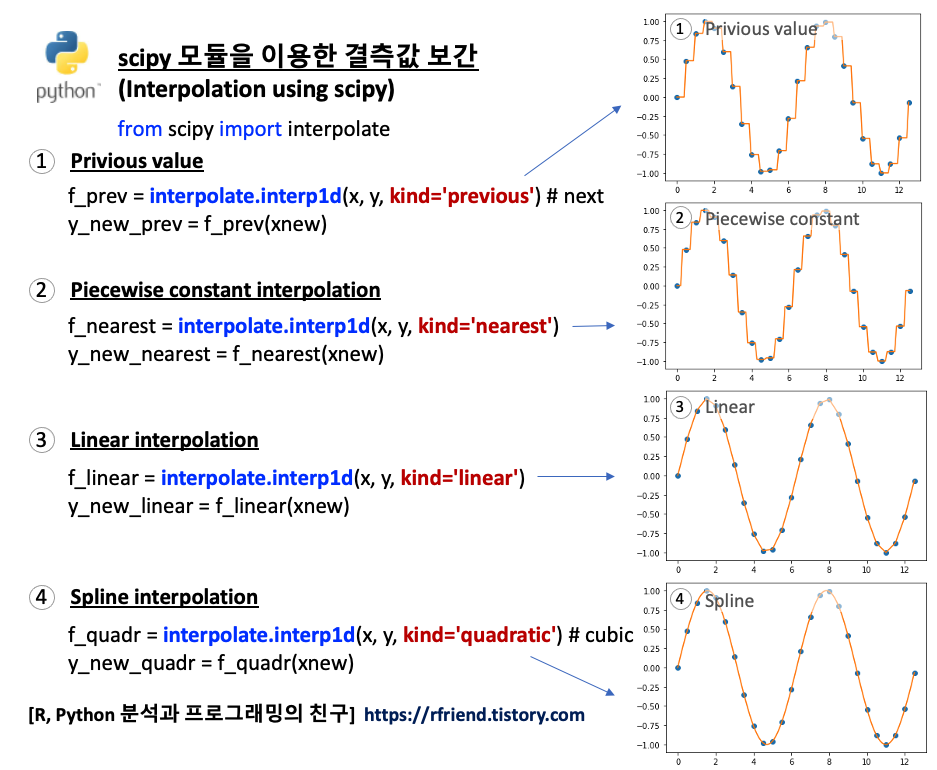
아래 코드는 Python으로 Lot, Cell, Parameter ID 별로 for loop 순환문을 사용해서 pandas 의 interpolate() 메소드를 사용해서 시계열 데이터의 결측값을 선형 보간(linear interpolation) 한 것입니다.
(forward fill 로 먼저 선형 보간을 해주고, 그 다음에 만약에 첫번째 행에 결측값이 있을 경우에 backward fill 로 이후 값과 같은 값으로 결측값을 채워줍니다.)
순차적으로 for loop 순환문을 돌기 때문에 시간이 오래 걸립니다.
#%% [Step 2] linear interpolation
from datetime import datetime
start_time = datetime.now()
## reading csv files in the base_dir
file_list = os.listdir(base_dir)
for file_nm in file_list:
# by Lot
if file_nm[-3:] == "csv":
# read csv file
ts_df = pd.read_csv(os.path.join(base_dir, file_nm))
# blank DataFrame for saving the interpolated time series later
ts_df_interpolated = pd.DataFrame()
# cell & param ID lists
cell_list = np.unique(ts_df['cell_id'])
param_list = np.unique(ts_df['param_id'])
# interpolation by lot, cell, and param IDs
for cell_id in cell_list:
for param_id in param_list:
ts_df_tmp = ts_df[(ts_df.cell_id == cell_id) & (ts_df.param_id == param_id)]
## interpolating the missing values for equaly spaced time series data
ts_df_tmp.sort_values(by='timestamp_id', ascending=True) # sorting by TimeStamp first
ts_df_interpolated_tmp = ts_df_tmp.interpolate(method='values') # linear interploation
ts_df_interpolated_tmp = ts_df_interpolated_tmp.fillna(method='bfill') # backward fill for the first missing row
ts_df_interpolated = pd.concat([ts_df_interpolated, ts_df_interpolated_tmp], axis=0)
# export DataFrame to local folder as a csv file
ts_df_interpolated.to_csv(os.path.join(interpolated_dir, file_nm), index=False)
print(file_nm, "is successfully interpolated.")
time_elapsed = datetime.now() - start_time
print("----------" * 5)
print("Time elapsed (hh:mm:ss.ms) {}".format(time_elapsed))
print("----------" * 5)
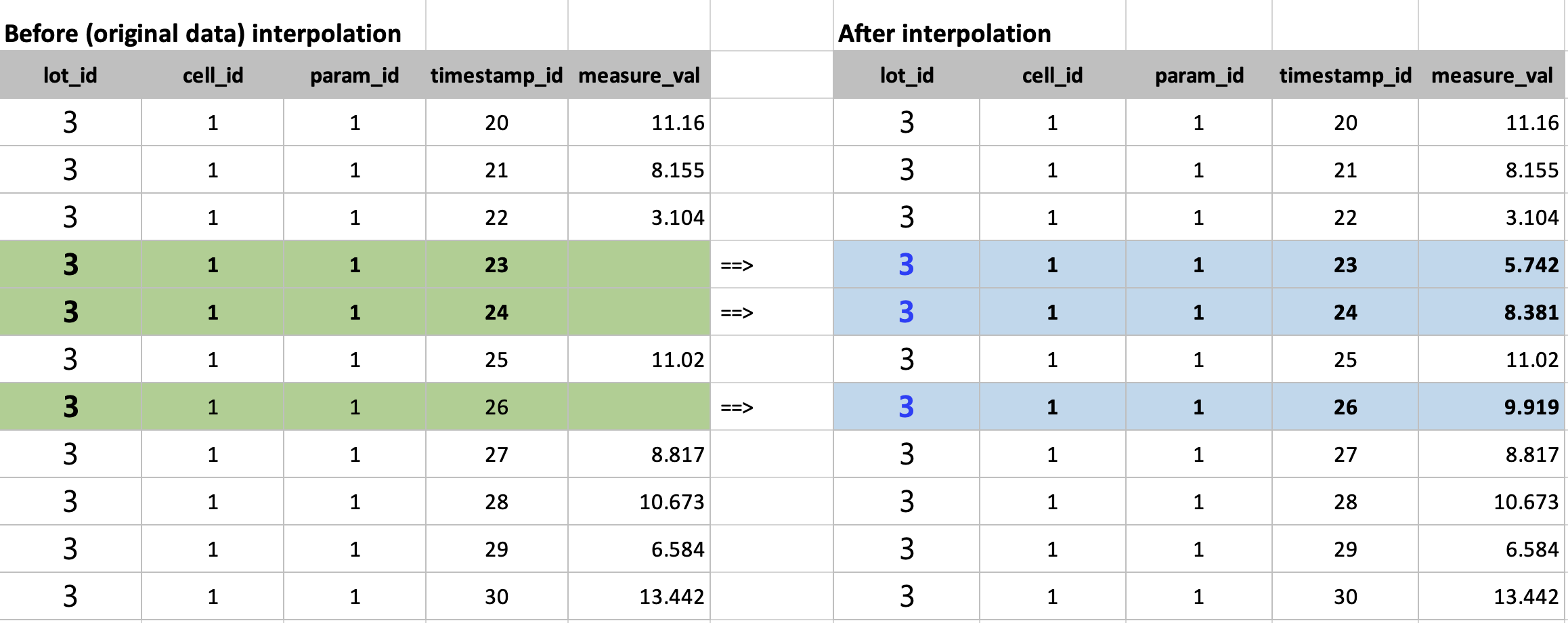
# # Before interplolation
# 3,1,1,20,11.160795506036791
# 3,1,1,21,8.155949904188175
# 3,1,1,22,3.1040644143505407
# 3,1,1,23, <-- missing
# 3,1,1,24, <-- missing
# 3,1,1,25,11.020504352275342
# 3,1,1,26, <-- missing
# 3,1,1,27,8.817922501760519
# 3,1,1,28,10.673174873272234
# 3,1,1,29,6.584669096660191
# 3,1,1,30,13.442427337943553
# # After interpolation
# 3,1,1,20,11.160795506036791
# 3,1,1,21,8.155949904188175
# 3,1,1,22,3.1040644143505407
# 3,1,1,23,5.742877726992141 <-- interpolated
# 3,1,1,24,8.381691039633742 <-- interpolated
# 3,1,1,25,11.020504352275342
# 3,1,1,26,9.919213427017931 <-- interpolated
# 3,1,1,27,8.81792250176052
# 3,1,1,28,10.673174873272234
# 3,1,1,29,6.584669096660191
# 3,1,1,30,13.442427337943554
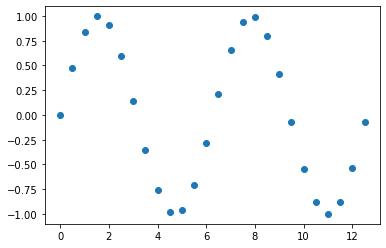
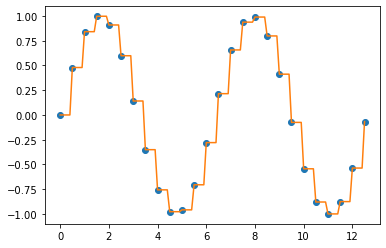
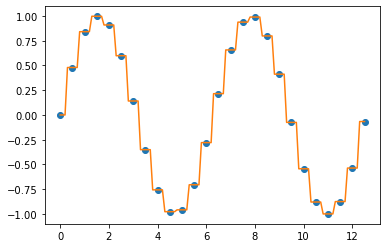
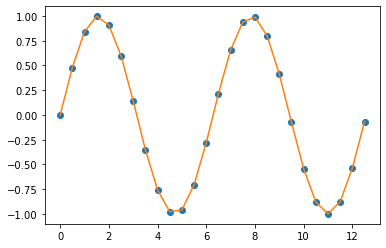
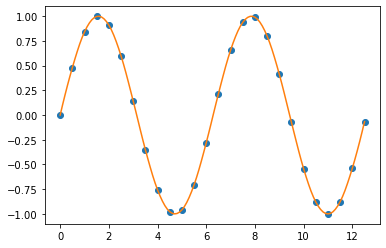
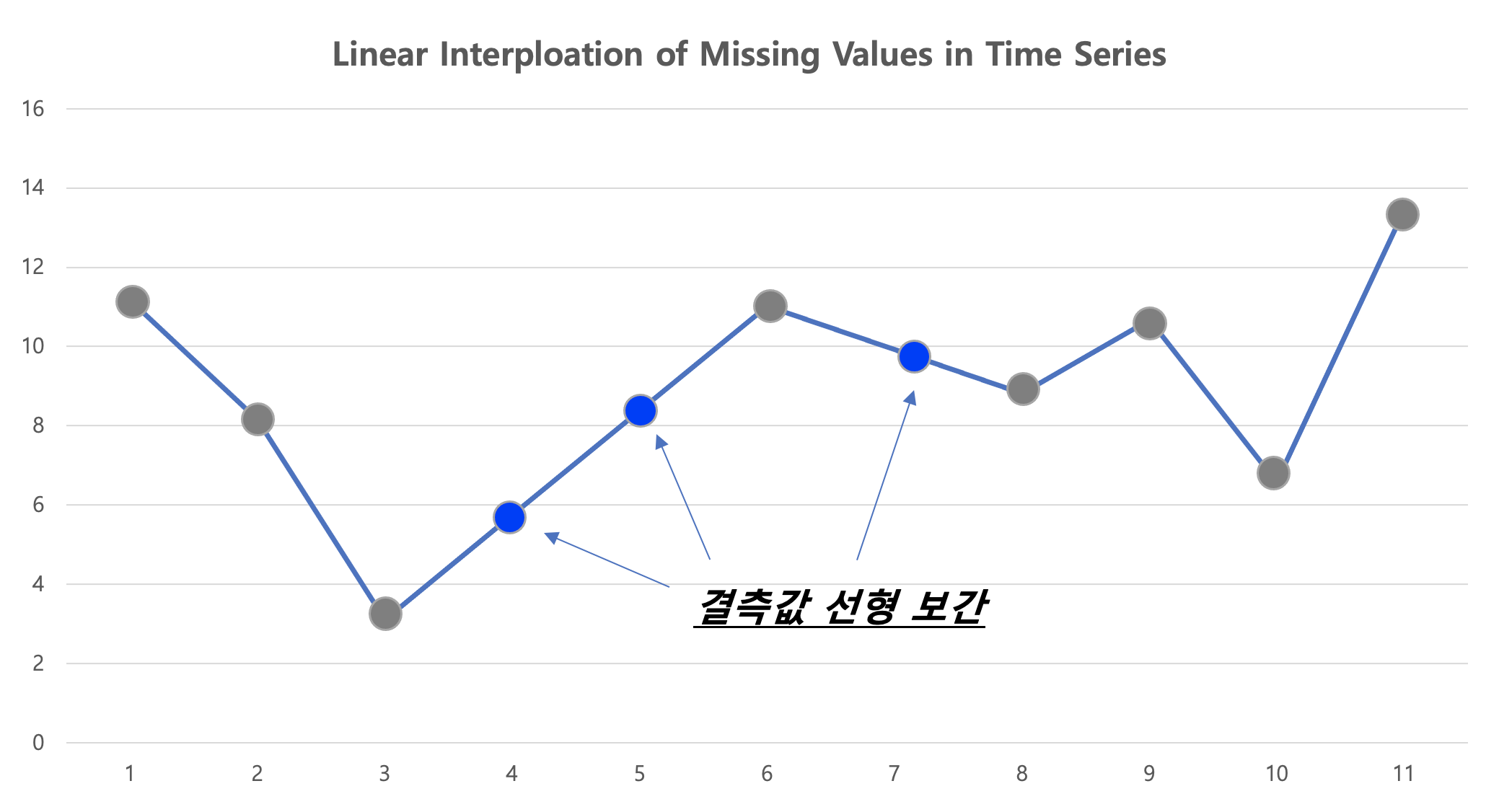
아래 화면캡쳐는 선형보간하기 전에 결측값이 있을 때와, 이를 선형보간으로 값을 생성한 후의 예시입니다.
아래 선 그래프의 파란색 점 부분이 원래 값에서는 결측값 이었던 것을 선형 보간(linear interpolation)으로 채워준 후의 모습입니다. 선형보간이므로 측정된 값으로 선형회귀식을 적합하고, 결측값 부분의 X 값을 입력해서 Y를 예측하는 방식으로 결측값을 보간합니다.
아래 코드는 데이터가 Greenplum DB에 적재되어 있다고 했을 때,
(2-1) Python으로 Greenplum DB에 access하여 데이터를 Query 해와서 pandas DataFrame으로 만들고
(2-2) Pytnon pandas 의 interpolate() 메소드를 사용해서 선형보간을 한 다음에
(2-3) 선형보간된 DataFrame을 pandas 의 to_sql() 메소드를 사용해서 다시 Greenplum DB에 적재
하는 코드입니다. 이를 for loop 순환문을 사용해서 Lot 의 개수만큼 실행시켜 주었습니다.
순차적으로 for loop 순환문을 돌기 때문에 시간이 오래 걸립니다.
#%% Greenplum credentials
user = 'username'
password = 'password'
host = 'ip_address'
port = 'port'
db = 'databasename'
connection_string = "postgresql://{user}:{password}@{host}:{port}/{db}".\
format(user=user,
password=password,
host=host,
port=port,
db=db)
#%%
# helper function: query to pandas DataFrame
def gpdb_query(query):
import psycopg2 as pg
import pandas as pd
conn = pg.connect(connection_string)
cursor = conn.cursor()
cursor.execute(query)
col_names = [desc[0] for desc in cursor.description]
result_df = pd.DataFrame(cursor.fetchall(), columns=col_names)
cursor.close()
conn.close()
return result_df
#%%
# UDF for running a query
def interpolator(lot_id):
#import pandas as pd
query = """
SELECT *
FROM ts_data
WHERE
lot_id = {lot_id}
""".format(
lot_id = lot_id)
ts_df = gpdb_query(query)
ts_df = ts_df.astype({
'measure_val': float
})
## interpolating the missing values for equaly spaced time series data
ts_df_interpolated = pd.DataFrame()
for cell_id in (np.arange(cell_num)+1):
for param_id in (np.arange(param_num)+1):
ts_df_tmp = ts_df[(ts_df.cell_id == cell_id) & (ts_df.param_id == param_id)]
ts_df_tmp.sort_values(by='timestamp_id', ascending=True) # sorting by TimeStamp first
ts_df_interpolated_tmp = ts_df_tmp.interpolate(method='values') # linear interploation
ts_df_interpolated_tmp = ts_df_interpolated_tmp.fillna(method='bfill') # backward fill for the first missing row
ts_df_interpolated = pd.concat([ts_df_interpolated, ts_df_interpolated_tmp], axis=0)
# export DataFrame to local folder as a csv file
#ts_df_interpolated.to_csv(os.path.join(interpolated_dir, file_nm), index=False)
#print(file_nm, "is successfully interpolated.")
return ts_df_interpolated
#%%
# UDF for importing pandas DataFrame to Greenplum DB
def gpdb_importer(lot_id, connection_string):
import sqlalchemy
from sqlalchemy import create_engine
engine = create_engine(connection_string)
# interpolation
ts_data_interpolated = interpolator(lot_id)
# inserting to Greenplum
ts_data_interpolated.to_sql(
name = 'ts_data_interpolated_python',
con = engine,
schema = 'equipment',
if_exists = 'append',
index = False,
dtype = {'lot_id': sqlalchemy.types.INTEGER(),
'cell_id': sqlalchemy.types.INTEGER(),
'param_id': sqlalchemy.types.INTEGER(),
'timestamp_id': sqlalchemy.types.INTEGER(),
'measure_val': sqlalchemy.types.Float(precision=6)
})
#%%
from datetime import datetime
start_time = datetime.now()
import pandas as pd
import os
import numpy as np
for lot_id in (np.arange(lot_num)+1):
gpdb_importer(lot_id, connection_string)
print("lot_id", lot_id, "is successfully interpolated.")
time_elapsed = datetime.now() - start_time
print("----------" * 5)
print("Time elapsed (hh:mm:ss.ms) {}".format(time_elapsed))
print("----------" * 5)
(3) Greenplum에서 PL/Python으로 분산병렬처리하여 시계열 데이터 결측값을 보간하기
(interpolation in parallel using PL/Python on Greenplum)
Greenplum에서 PL/Python으로 병렬처리할 때는 (a) 사용자 정의 함수(UDF) 정의, (b) 사용자 정의 함수 실행의 두 단계를 거칩니다.
Greenplum DB에서 PL/Python으로 분산병렬처리를 하면 위의 (2)번에서 Python으로 for loop 순환문으로 순차처리한 것 대비 Greenplum DB 내 노드의 개수에 비례하여 처리 속도가 줄어들게 됩니다. (가령, 노드가 8개이면 병렬처리의 총 처리 소요시간은 순차처리했을 때의 총 소요시간의 1/8 로 줄어듭니다.)

(3-1) PL/Python 으로 시계열 데이터 결측값을 선형보간하는 사용자 정의함수 정의 (define a UDF)
-- defining the PL/Python UDF
DROP FUNCTION IF EXISTS plpy_interp(numeric[]);
CREATE OR REPLACE FUNCTION plpy_interp(measure_val_arr numeric[])
RETURNS numeric[]
AS $$
import numpy as np
import pandas as pd
measure_val = np.array(measure_val_arr, dtype='float')
ts_df = pd.DataFrame({
'measure_val': measure_val
})
# interpolation by lot, cell, and param IDs
ts_df_interpolated = ts_df.interpolate(method='values') # linear interploation
ts_df_interpolated = ts_df_interpolated.fillna(method='bfill') # backward fill for the first missing row
return ts_df_interpolated['measure_val']
$$ LANGUAGE 'plpythonu';
(3-2) 위에서 정의한 시계열 데이터 결측값을 선형보간하는 PL/Python 사용자 정의함수 실행
PL/Python의 input 으로는 SQL의 array_agg() 함수를 사용해서 만든 Array 데이터를 사용하며, PL/Python에서는 SQL의 Array를 Python의 List 로 변환(converting) 합니다.
-- array aggregation as an input
DROP TABLE IF EXISTS tab1;
CREATE TEMPORARY TABLE tab1 AS
SELECT
lot_id
, cell_id
, param_id
, ARRAY_AGG(timestamp_id ORDER BY timestamp_id) AS timestamp_id_arr
, ARRAY_AGG(measure_val ORDER BY timestamp_id) AS measure_val_arr
FROM ts_data
GROUP BY lot_id, cell_id, param_id
DISTRIBUTED RANDOMLY ;
ANALYZE tab1;
-- executing the PL/Python UDF
DROP TABLE IF EXISTS ts_data_interpolated;
CREATE TABLE ts_data_interpolated AS (
SELECT
lot_id
, cell_id
, param_id
, timestamp_id_arr
, plpy_interp(measure_val_arr) AS measure_val_arr -- plpython UDF
FROM tab1 AS a
) DISTRIBUTED BY (lot_id);
아래 코드는 numeric array 형태로 반환한 선형보간 후의 데이터를 unnest() 함수를 사용해서 보기에 편하도록 long format 으로 풀어준 것입니다.
-- display the interpolated result
SELECT
lot_id
, cell_id
, param_id
, UNNEST(timestamp_id_arr) AS timestamp_id
, UNNEST(measure_val_arr) AS measure_val
FROM ts_data_interpolated
WHERE lot_id = 1 AND cell_id = 1 AND param_id = 1
ORDER BY lot_id, cell_id, param_id, timestamp_id
LIMIT 100;
결측값을 포함하고 있는 원래 데이터셋을 아래 SQL query 로 조회해서, 위의 선형보간 된 후의 데이터셋과 비교해볼 수 있습니다.
-- original dataset with missing value
SELECT
lot_id
, cell_id
, param_id
, timestamp_id
, measure_val
FROM ts_data
WHERE lot_id = 1 AND cell_id = 1 AND param_id = 1
ORDER BY lot_id, cell_id, param_id, timestamp_id
LIMIT 100;
이번 포스팅이 많은 도움이 되었기를 바랍니다.
행복한 데이터 과학자 되세요! :-)

'Greenplum and PostgreSQL Database' 카테고리의 다른 글
| [PostgreSQL, Greenplum] SQL Query 실행 순서 (SQL order of execution) (0) | 2023.03.26 |
|---|---|
| [PostgreSQL, Greenplum] 차원이 다른 Array를 2D Array로 aggregation 하기 (0) | 2023.03.05 |
| [PostgreSQL, Greenplum] 스펙트럼 분석 PL/Python을 활용한 병렬처리 (1) | 2022.10.23 |
| [PostgreSQL/ Greenplum] 정규분포에서 난수를 생성하여 샘플 테이블 만들기 (0) | 2022.09.04 |
| [PostgreSQL, Greenplum] 여러개의 문자열 매칭 SQL (0) | 2022.06.26 |