폴더에 자잘하게 쪼개진 여러개의 파일들이 있을 때, 그리고 이 파일들을 일일이 R로 불러오기 해야 할 때, 더그리고 이들 불러온 파일을 한개의 데이터셋을 합쳐야 할 때 (이쪽 동네 전문용어로) 노가다를 하지 않고 좀더 스마트하게 하는 방법을 소개하겠습니다.
순서는 다음과 같습니다.
- (1) 폴더 경로 객체로 만들기
- (2) 폴더 내 파일들 이름을 list-up 하여 객체로 만들기
- (3) 파일 개수 객체로 만들기
- (4) 폴더 내 파일들을 LOOP 돌려서 불러오기 : read.table()
- (5) 파일을 내보내면서 합치기 : write.table(dataset, APPEND = TRUE)
- (6) 데이터프레임으로 불러오기, 칼럼 이름 넣기 : read.table(dataset_all, col.names = c())
자, 예를 들면서 순서대로 R script 설명하겠습니다.
아래의 화면캡쳐 예시처럼 MyDocuments > R > FILES 폴더 아래에 daily로 쪼개진 10개의 text 파일들이 들어있다고 해봅시다. (10개 정도야 일일이 불어올 수도 있겠지만, 100개, 1,000개 파일이 들어있다면?)
- (1) 폴더 경로 객체로 만들기
## cleaning up environment
rm(list=ls())
## making directory as an object
src_dir <- c("C:/Users/Owner/Documents/R/FILES") # 경로 구분 : '\'를 '/'로 바꿔야 함
src_dir
#[1] "C:/Users/Owner/Documents/R/FILES"
- (2) 폴더 내 파일들 이름을 list-up 하여 객체로 만들기 : list.files()
# listing up name of files in the directory => object
src_file <- list.files(src_dir) # list
src_file
#[1] "day_20160701.txt" "day_20160702.txt" "day_20160703.txt" "day_20160704.txt"
#[5] "day_20160705.txt" "day_20160706.txt" "day_20160707.txt" "day_20160708.txt"
#[9] "day_20160709.txt" "day_20160710.txt"
"C:/Users/Owner/Documents/R/FILES" 디렉토리에 들어있는 파일들을 열어보면 아래와 같은 데이터들이 들어있습니다. (가상으로 만들어 본 것임) daily로 집계한 데이터들이 들어있네요.
- (3) 파일 개수 객체로 만들기 : length(list)
# counting number of files in the directory => object
src_file_cnt <- length(src_file)
src_file_cnt
#[1] 10
여기까지 R을 실행하면 아래와 같이 environment 창에 객체들이 생겼음을 확인할 수 있습니다.
- (4) 폴더 내 파일들을 LOOP 돌려서 불러오기
=> (5) 파일을 내보내면서 합치기 : write.table(dataset, APPEND = TRUE)
: for(i in 1:src_file_cnt) {read.table()
write.table(dataset, append = TRUE)}
## write.table one by one automatiically, using loop program
for(i in 1:src_file_cnt) {
# write.table one by one automatiically, using loop program
dataset <- read.table(
paste(src_dir, "/", src_file[i], sep=""),
sep=",",
header=F,
stringsAsFactors = F)
# dataset exporting with 'APPEND = TREU' option, filename = dataset_all.txt
write.table(dataset,
paste(src_dir, "/", "dataset_all.txt", sep=""),
sep = ",",
row.names = FALSE,
col.names = FALSE,
quote = FALSE,
append = TRUE) # appending dataset (stacking)
# delete seperate datasets
rm(dataset)
# printing loop sequence at console to check loop status
print(i)
}
#[1] 1
#[1] 2
#[1] 3
#[1] 4
#[1] 5
#[1] 6
#[1] 7
#[1] 8
#[1] 9
#[1] 10
여기까지 실행을 하면 아래처럼 MyDocuments>R>FILES 폴더 아래에 'dataset_all.txt' 라는 새로운 텍스트 파일이 하나 생겼음을 확인할 수 있습니다.
새로 생긴 'dataset_all.txt' 파일을 클릭해서 열어보면 아래와 같이 'day_20160701.txt' ~ 'day_20160710.txt'까지 10개 파일에 흩어져있던 데이터들이 차곡차곡 쌓여서 합쳐져 있음을 확인할 수 있습니다.
- (6) 데이터 프레임으로 불러오기 : read.table()
칼럼 이름 붙이기 : col.names = c("var1", "var2", ...)
# reading dataset_all with column names
dataset_all_df <- read.table(
paste(src_dir, "/", "dataset_all.txt", sep=""),
sep = ",",
header = FALSE, # no column name in the dataset
col.names = c("ymd", "var1", "var2", "var3", "var4", "var5", + "var6", "var7", "var8", "var9", "var10"), # input column names
stringsAsFactor = FALSE,
na.strings = "NA") # missing value : "NA"
우측 상단의 environment 창에서 'dataset_all_df' 데이터 프레임이 새로 생겼습니다.
클릭해서 열어보면 아래와 같이 'day_20160701.txt' ~ 'day_20160710.txt'까지 데이터셋이 합쳐져있고, "ymd", "var1" ~ "var10" 까지 칼럼 이름도 생겼습니다.
프로그래밍을 통한 자동화가 중요한 이유, 우리의 시간은 소중하니깐요~! ^^
이번 포스팅이 도움이 되었다면 아래의 '공감 ~♡'를 꾸욱 눌러주세요.
====================================================================
(2018.03.14일 내용 추가)
댓글 질문에 '폴더에 있는 개별 파일을 하나씩 읽어와서 하나씩 DataFrame 객체로 메모리상에 생성하는 방법에 대한 질문이 있어서 코드 추가해서 올립니다. 위에서 소개한 방법과 전반부는 동일하구요, 마지막에 루프 돌릴 때 assign() 함수로 파일 이름을 할당하는 부분만 조금 다릅니다.
| #========================================================= # read all files in a folder and make a separate dataframe #========================================================= rm(list=ls()) # clear all # (1) directory src_dir <- c("D:/admin/Documents/R/R_Blog/326_read_all_files") # (2) make a file list of all files in the folder src_file <- list.files(src_dir) src_file  # (3) count the number of files in the directory => object src_file_cnt <- length(src_file) src_file_cnt # 5 # (4) read files one by one using looping # => make a dataframe one by one using assign function for (i in 1:src_file_cnt){ assign(paste0("day_", i), read.table(paste0(src_dir, "/", src_file[i]), sep = ",", header = FALSE)) print(i) # check progress } rm(src_dir, src_file, src_file_cnt, i) # delete temp objects ls() # list-up all dataframes  |
==================================================
(2021.08.24일 추가)
댓글에 "여러개의 파일을 하나로 합칠 때 "파일 이름을 데이터 프레임의 새로운 칼럼에 값으로 추가한 후"에 합치는 방법"에 대한 문의가 있었습니다. 댓글란에 코드 블락을 복사해 넣으면 들여쓰기가 무시되어서 보기가 힘들므로 본문에 예제 코드 추가해 놓습니다.
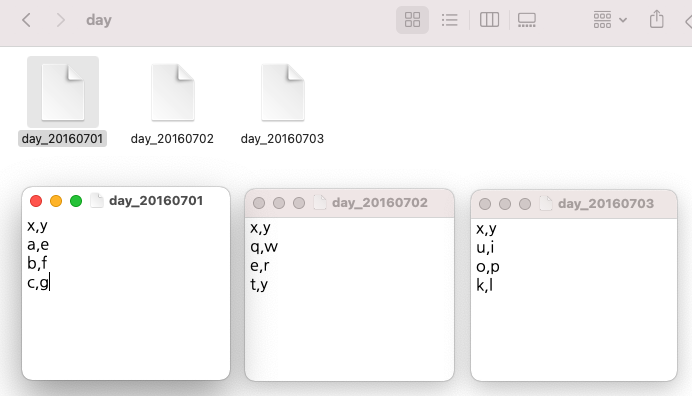
간단한 샘플 텍스트 파일 3개 만들어서 for loop 순환문으로 각 파일 읽어온 후, 파일 이름을 새로운 칼람 'z'의 값으로 할당 해주고, blank data.frame 인 'day_all' 에 순차적으로 rbind 해주었습니다.
##--------------------------------------------------------
## add new column with file name and append all dataframes
##--------------------------------------------------------
## blank data.frame to save all files later
day_all <- data.frame()
## file list
src_dir <- c("/Users/lhongdon/Documents/day")
src_file <- list.files(src_dir)
src_file
# [1] "day_20160701" "day_20160702" "day_20160703"
for (i in 1:length(src_file)){
# read dataset 1 by 1 sequentially
day_temp <- read.table(
paste0(src_dir, "/", src_file[i]),
sep=",",
header=T,
stringsAsFactors=F)
# add filename as a new column
day_temp$z <- src_file[i]
# rbind day_temp to day_all data.frame
day_all <- rbind(day_all, day_temp)
#print(i) # for progress check
}
print(day_all)
# x y z
# 1 a e day_20160701
# 2 b f day_20160701
# 3 c g day_20160701
# 4 q w day_20160702
# 5 e r day_20160702
# 6 t y day_20160702
# 7 u i day_20160703
# 8 o p day_20160703
# 9 k l day_20160703
=============================
(2021.08.25 일 추가)
댓글에 추가 질문이 달려서 요건에 맞게 코드를 더 추가하였습니다.
중첩 for loop 문에 조건절이 여러개 들어가다 보니 코드가 많이 복잡해졌네요.
[데이터 전처리 요건 ]
1. 로컬 머신 폴더 내 여러개의 csv 파일을 읽어와서 한개의 R data.frame 으로 통합
2. 이때 개별 csv 파일로 부터 읽어들인 데이터를 특정 개수의 [행 * 열] data.frame 으로 표준화
- 가령, 3 행 (rows) * 3 열 (columns) 의 data.frame 으로 표준화하기 원한다면
- 개별 csv 파일로 부터 읽어들인 데이터의 행(row)의 개수가 3보다 크면 1~3행까지만 가져와서 합치고 나머지는 버림. 반대로 3개 행보다 부족하면 'NA' 결측값으로 처리함.
- 개별 csv 파일로 부터 읽어들인 데이터의 열(column)이 타켓 칼럼 이름(가령, "x", "y", "z") 중에서 특정 칼럼이 없다면 그 칼럼의 값은 모두 'NA' 결측값으로 처리함.(가령, csv 파일 내에 "x", "y" 만 있고 "z" 칼럼은 없다면 "z" 칼럼을 만들어주고 대신 값은 모두 'NA' 처리해줌)
3. 'day' 라는 칼럼을 새로 만들어서 파일 이름(day 날짜가 들어가 있음)을 값으로 넣어줌
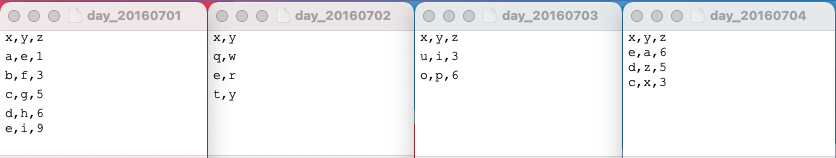
[ 예제 데이터 ]
##--------------------------------------------------------
## (1) 3 rows & 3 cols DataFrame
## (2) add new column with file name and append all dataframes
##--------------------------------------------------------
## blank data.frame to save all files later
day_all <- data.frame()
## file list
src_dir <- c("/Users/lhongdon/Documents/day")
src_file <- list.files(src_dir)
src_file
# [1] "day_20160701" "day_20160702" "day_20160703" "day_20160704"
## setting target rows & cols
row_num <- 3 # set your target number of rows
col_name <- c("x", "y", "z") # set your target name of columns
for (i in 1:length(src_file)){
# read dataset 1 by 1 sequentially
day_temp <- read.table(
paste0(src_dir, "/", src_file[i]),
sep=",",
header=T,
stringsAsFactors=F)
##-- if the number of rows is less than 3 then 'NA',
##-- if the number of rows is greater than 3 than ignore them
##-- if the name of columns is not in col_nm then 'NA'
# blank temp dataframe with 3 rows and 3 columns
tmp_r3_c3 <- data.frame(matrix(rep(NA, row_num*col_num),
nrow=row_num,
byrow=T))
names(tmp_r3_c3) <- col_name
tmp_row_num <- nrow(day_temp)
tmp_col_name <- colnames(day_temp)
r <- ifelse(row_num > tmp_row_num, tmp_row_num, row_num)
for (j in 1:r) {
for (k in 1:length(tmp_col_name)) {
tmp_r3_c3[j, tmp_col_name[k]] <- day_temp[j, tmp_col_name[k]]
}
}
# add filename as a new column 'day'
tmp_r3_c3$day <- src_file[i]
# rbind day_temp to day_all data.frame
day_all <- rbind(day_all, tmp_r3_c3)
rm(tmp_r3_c3)
print(i) # for progress check
}
print(day_all)
# x y z day
# 1 a e 1 day_20160701
# 2 b f 3 day_20160701
# 3 c g 5 day_20160701
# 4 q w NA day_20160702
# 5 e r NA day_20160702
# 6 t y NA day_20160702
# 7 u i 3 day_20160703
# 8 o p 6 day_20160703
# 9 <NA> <NA> NA day_20160703
# 10 e a 6 day_20160704
# 11 d z 5 day_20160704
# 12 c x 3 day_20160704
많은 도움이 되었기를 바랍니다.
행복한 데이터 과학자 되세요! :-)
'R 분석과 프로그래밍 > R 프로그래밍' 카테고리의 다른 글
| [R] 데이터프레임 다수 item을 가진 문자형 변수를 분리 후에 세로로 재구조화하기 (11) | 2016.09.29 |
|---|---|
| [R] 여러개의 데이터프레임을 한꺼번에 하나의 데이터프레임으로 묶기, data.table package : rbindlist(data) (8) | 2016.07.10 |
| R "target of assignment expands to non-language object" error : assign() (2) | 2015.09.22 |
| ggplot2 저장 : ggsave(), console 내용 text 저장 : capture.output() (0) | 2015.09.17 |
| R 사용자 정의 함수 (User Defined Function) (4) | 2015.09.11 |










