이번 포스팅에서는 Python을 사용해서 두 집단 간 평균이 같은지 아니면 다른지를 검정하는 t-test 를 해보겠습니다.
연속형 확률분포인 t-분포 (Student's t-distribution) 에 대해서는 https://rfriend.tistory.com/110 를 참고하세요.
R을 사용한 독립된 두 집단간 모평균 차이에 대한 검정은 https://rfriend.tistory.com/127 를 참고하세요.
모집단의 평균과 분산에 대해서는 알지 못하는 경우가 많으므로, 보통은 모집단에서 무작위로 표본을 추출(random sampling)해서 모집단의 평균과 분산을 추정합니다. 표본의 크기가 작은 집단 간 평균의 차이가 있는지를 검정할 때 t-분포에 기반한 t-통계량(t-statistics)을 사용하여 검정을 합니다.
t-검정은 대상 표본 집단이 1개인지 2개인지에 따라서 아래와 같이 구분할 수 있습니다.
* One-sample t-test : 모집단의 평균이 귀무가설의 특정 평균 값과 같은지를 검정
* Two-sample t-test: 두 모집단의 평균이 같다는 귀무가설을 검정
One-sample t-test와 Two-sample t-test에서 사용하는 통계량에 대해서는 아래에 정리해보았습니다.

여기서부터는 독립된 두 표본 간의 평균 차이에 대한 t-검정 (independent two-sample t-test) 에 대해서만 자세하게 소개하도록 하겠습니다.
(1) Two-sample t-test 의 가설 (Hypothesis)
- 귀무가설 (Null Hypothesis, H0): Mu1 = M2 (두 모집단의 평균이 같다)
- 대립가설 (Alternative Hypothesis, H1)
-. 양측검정 대립가설 (two-sided test H1): Mu1 ≠ Mu2 (두 모집단의 평균이 같지 않다)
-. 우측검정 대립가설 (right-tailed test H1): Mu1 > M2 (모집단1의 평균이 모집단2의 평균보다 크다)
-. 좌측검정 대립가설 (left-tailed test H1): M1 < M2 (모집단1의 평균이 모집단2의 평균보다 작다)
t-test 를 통해 나온 p-value 가 유의수준보다 작으면 귀모가설을 기각하고 대립가설을 채택(즉, 두 모집단의 평균이 차이가 있다)하게 됩니다.
(2) Two-sample t-test 의 가정사항 (Assumptions)
Two-sample t-test 의 결과가 유효하기 위해서는 아래의 가정사항을 충족시켜야 합니다.
(a) 한 표본의 관측치는 다른 표본의 관측치와 독립이다. (independent)
(b) 데이터는 정규분포를 따른다. (normally distributed)
(c) 두 집단의 표본은 동일한 분산을 가진다. (the same variance).
(--> 이 가설을 만족하지 못하면 Welch's t-test 를 실행합니다.)
(d) 두 집단의 표본은 무작위 표본추출법을 사용합니다. (random sampling)
정규성 검정(normality test)을 위해서 Kolmogorov-Smirnov test, Shapiro-Wilk test, Anderson-Darling test 등을 사용합니다. 등분산성 검정(Equal-Variance test) 을 위해서 Bartlett test, Fligner test, Levene test 등을 사용합니다.
(3) Python을 이용한 Two-sample t-test 실행
(3-1) 샘플 데이터 생성
먼저 numpy 모듈을 사용해서 정규분포로 부터 각 관측치 30개를 가지는 표본을 3개 무작위 추출해보겠습니다. 이중 표본집단 2개는 평균과 분산이 동일한 정규분포로 부터 무작위 추출하였으며, 나머지 1개 집단은 평균이 다른 정규분포로 부터 무작위 추출하였습니다.
## generating sample dataset
import numpy as np
np.random.seed(1004) # for reproducibility
x1 = np.random.normal(loc=0, scale=1, size=30) # the same mean
x2 = np.random.normal(loc=0, scale=1, size=30) # the same mean
x3 = np.random.normal(loc=4, scale=1, size=30) # different mean
x1
# array([ 0.59440307, 0.40260871, -0.80516223, 0.1151257 , -0.75306522,
# -0.7841178 , 1.46157577, 1.57607553, -0.17131776, -0.91448182,
# 0.86013945, 0.35880192, 1.72965706, -0.49764822, 1.7618699 ,
# 0.16901308, -1.08523701, -0.01065175, 1.11579838, -1.26497153,
# -1.02072516, -0.71342119, 0.57412224, -0.45455422, -1.15656742,
# 1.29721355, -1.3833716 , 0.3205909 , -0.59086187, -1.43420648])
x2
# array([ 0.60998011, 0.51266756, 1.9965168 , 1.42945668, 1.82880165,
# -1.40997132, 0.49433367, 0.9482873 , -0.35274099, -0.15359935,
# -1.18356064, -0.75440273, -0.85981073, 1.14256322, -2.21331694,
# 0.90651805, 2.23629 , 1.00743665, 1.30584548, 0.46669171,
# -0.49206651, -0.08727244, -0.34919043, -1.11363541, -1.71982966,
# -0.14033817, 0.90928317, -0.60012686, 1.03906073, -0.03332287])
x3
# array([2.96575604, 4.15929405, 4.33053582, 4.02563551, 3.90786096,
# 3.08148823, 4.3099129 , 2.75788362, 3.66886973, 2.35913334,
# 3.72460166, 3.94510997, 5.50604364, 2.62243844, 2.74438348,
# 4.16120867, 3.57878295, 4.2341905 , 2.79844805, 5.48131392,
# 4.29105321, 4.4022031 , 3.58533963, 5.00502917, 5.45376705,
# 3.92961847, 4.52897801, 1.62104705, 3.24945253, 5.10641762])
## Box plot
import matplotlib.pyplot as plt
import seaborn as sns
plt.figure(figsize=(10, 8))
sns.boxplot(data=[x1, x2, x3])
plt.xlabel("Group", fontsize=16)
plt.ylabel("Value", fontsize=16)
plt.xticks([0, 1, 2], ["x1", "x2", "x3"], fontsize=14)
plt.show()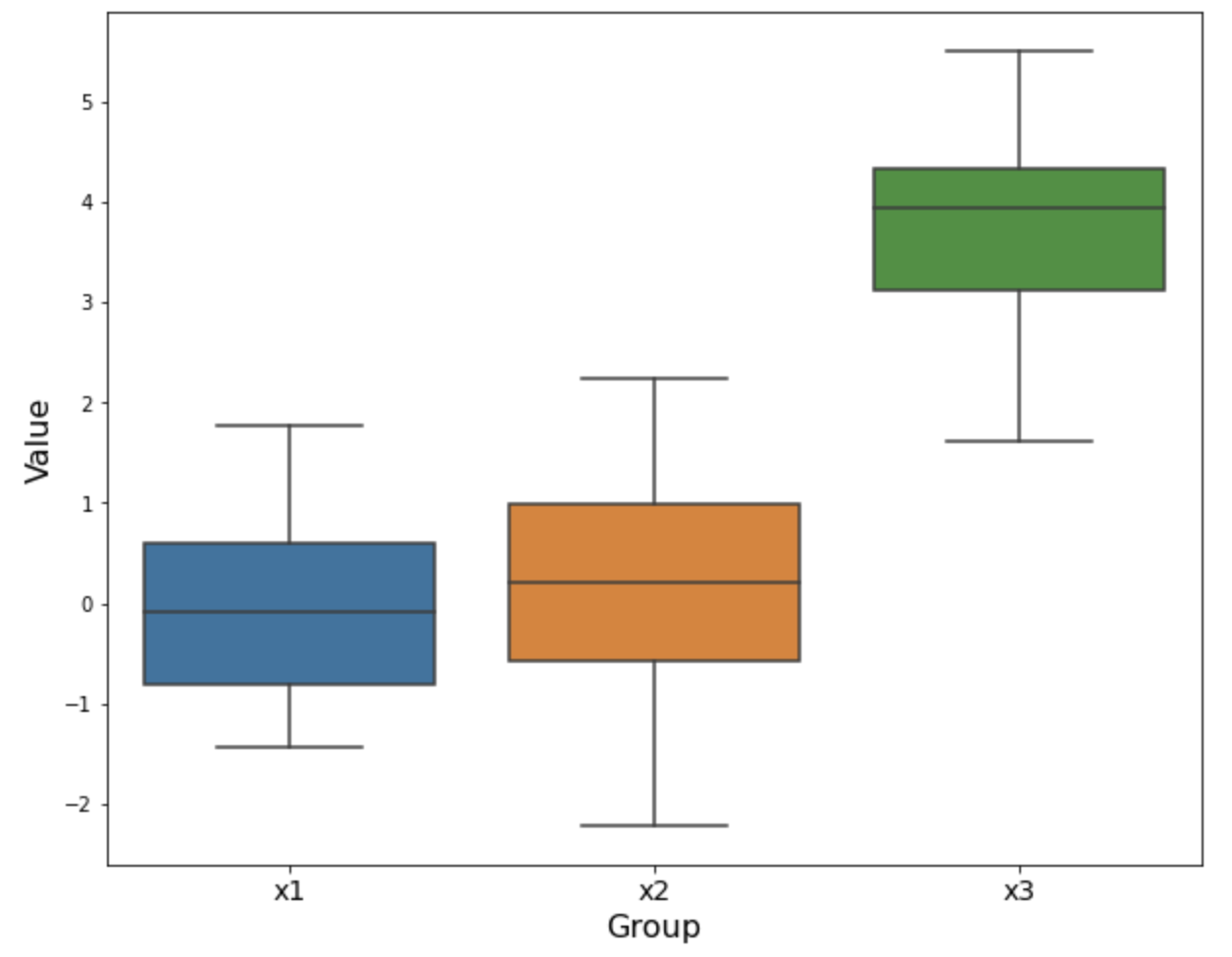
(3-2) t-test 가설 충족 여부 검정
t-검정의 가정사항으로서 정규성 검정(normality test)과 등분산성 검정 (equal variance test) 을 Python의 scipy 모듈을 사용해서 수행해보겠습니다.
* Kolmogorov-Smirnov Test 정규성 검정
- (귀무가설, H0): 집단의 데이터가 정규 분포를 따른다.
- (대립가설, H1): 집단의 데이터가 정규 분포를 따르지 않는다.
아래에 x1, x2, x3 의 세 집단에 대한 K-S 정규성 검정 결과 p-value 가 모두 유의수준 0.05 보다 크므로 귀무가설을 채택하여 세 집단의 데이터가 정규 분포를 따른다고 볼 수 있습니다.
## (1) Normality test using Kolmogorov-Smirnov Test
import scipy.stats as stats
t_stat_x1, p_val_x1 = stats.kstest(x1, 'norm', args=(x1.mean(), x1.var()**0.5))
t_stat_x2, p_val_x2 = stats.kstest(x2, 'norm', args=(x2.mean(), x2.var()**0.5))
t_stat_x3, p_val_x3 = stats.kstest(x3, 'norm', args=(x3.mean(), x3.var()**0.5))
print('[x1] t-statistics:', t_stat_x1, ' p-value:', p_val_x1)
print('[x2] t-statistics:', t_stat_x2, ' p-value:', p_val_x2)
print('[x3] t-statistics:', t_stat_x3, ' p-value:', p_val_x3)
# [x1] t-statistics: 0.13719205314969185 p-value: 0.577558008887932
# [x2] t-statistics: 0.11086245840821829 p-value: 0.8156064477001308
# [x3] t-statistics: 0.09056001868899977 p-value: 0.9477307432911599
다음으로 집단 x1과 x2, 집단 x1과 x3에 대한 등분산 가정 검정 결과, p-value 가 모두 유의수준 0.05 보다 크므로 두 집단 간 분산이 같다고 할 수 있습니다. (귀무가설 H0: 두 집단 간 분산이 같다.)
## (2) Equal variance test using Bartlett's tes
var_test_stat_x1x2, var_test_p_val_x1x2 = stats.bartlett(x1, x2)
var_test_stat_x1x3, var_test_p_val_x1x3 = stats.bartlett(x1, x3)
print('[x1 vs. x2]', 'statistic:', var_test_stat_x1x2, ' p-value:', var_test_p_val_x1x2)
print('[x1 vs. x3]', 'statistic:', var_test_stat_x1x3, ' p-value:', var_test_p_val_x1x3)
# [x1 vs. x2] statistic: 0.4546474955289549 p-value: 0.5001361557169177
# [x1 vs. x3] statistic: 0.029962346601998174 p-value: 0.8625756934286083
처음에 샘플 데이터를 생성할 때 정규분포로 부터 분산을 동일하게 했었으므로 예상한 결과대로 잘 나왔네요.
(3-3) 독립된 두 표본에 대한 t-test 평균 동질성 여부 검정
이제 독립된 두 표본에 대해 t-test 를 실행해서 두 표본의 평균이 같은지 다른지 검정을 해보겠습니다.
- (귀무가설 H0) Mu1 = Mu2 (두 집단의 평균이 같다)
- (대립가설 H1) Mu1 ≠ Mu2 (두 집단의 평균이 다르다)
분산은 서로 같으므로 equal_var = True 라고 매개변수를 설정해주었습니다.
그리고 양측검정(two-sided test) 을 할 것이므로 alternative='two-sided' 를 설정(default)해주면 됩니다. (왜그런지 자꾸 에러가 나서 일단 코멘트 부호 # 로 막아놨어요. scipy 버전 문제인거 같은데요... 흠... 'two-sided'가 default 설정이므로 # 로 막아놔도 문제는 없습니다.)
## (3) Identification test using Independent 2 sample t-test
## x1 vs. x2
import scipy.stats as stats
t_stat, p_val = stats.ttest_ind(x1, x2,
#alternative='two-sided', #‘less’, ‘greater’
equal_var=True)
print('t-statistic:', t_stat, ' p-value:', p_val)
#t-statistic: -0.737991822907993 p-value: 0.46349499774375136
#==> equal mean
## x1 vs. x3
import scipy.stats as stats
t_stat, p_val = stats.ttest_ind(x1, x3,
#alternative='two-sided', #‘less’, ‘greater’
equal_var=True)
print('t-statistic:', t_stat, ' p-value:', p_val)
#t-statistic: -15.34800563666855 p-value: 4.370531118607397e-22
#==> different mean
(3-1)에서 샘플 데이터를 만들 때 x1, x2 는 동일한 평균과 분산의 정규분포에서 무작위 추출을 하였으며, x3만 평균이 다른 정규분포에서 무작위 추출을 하였습니다.
위의 (3-3) t-test 결과를 보면 x1, x2 간 t-test 에서는 p-value 가 0.46으로서 유의수준 0.05 하에서 귀무가설(H0)을 채택하여 두 집단 x1, x2 의 평균은 같다고 판단할 수 있습니다.
x1, x3 집단 간 t-test 결과를 보면 p-value 가 4.37e-22 로서 유의수준 0.05 하에서 귀무가설(H0)을 기각(reject)하고 대립가설(H1)을 채택(accept)하여 x1, x3 의 평균이 다르다고 판단할 수 있습니다.
[ Reference ]
* Wikipedia Student's t-test: https://en.wikipedia.org/wiki/Student%27s_t-test
* Python scipy.stats.ttest_ind 메소드
: https://docs.scipy.org/doc/scipy/reference/generated/scipy.stats.ttest_ind.html
이번 포스팅이 많은 도움이 되었기를 바랍니다.
행복한 데이터 과학자 되세요! :-)




