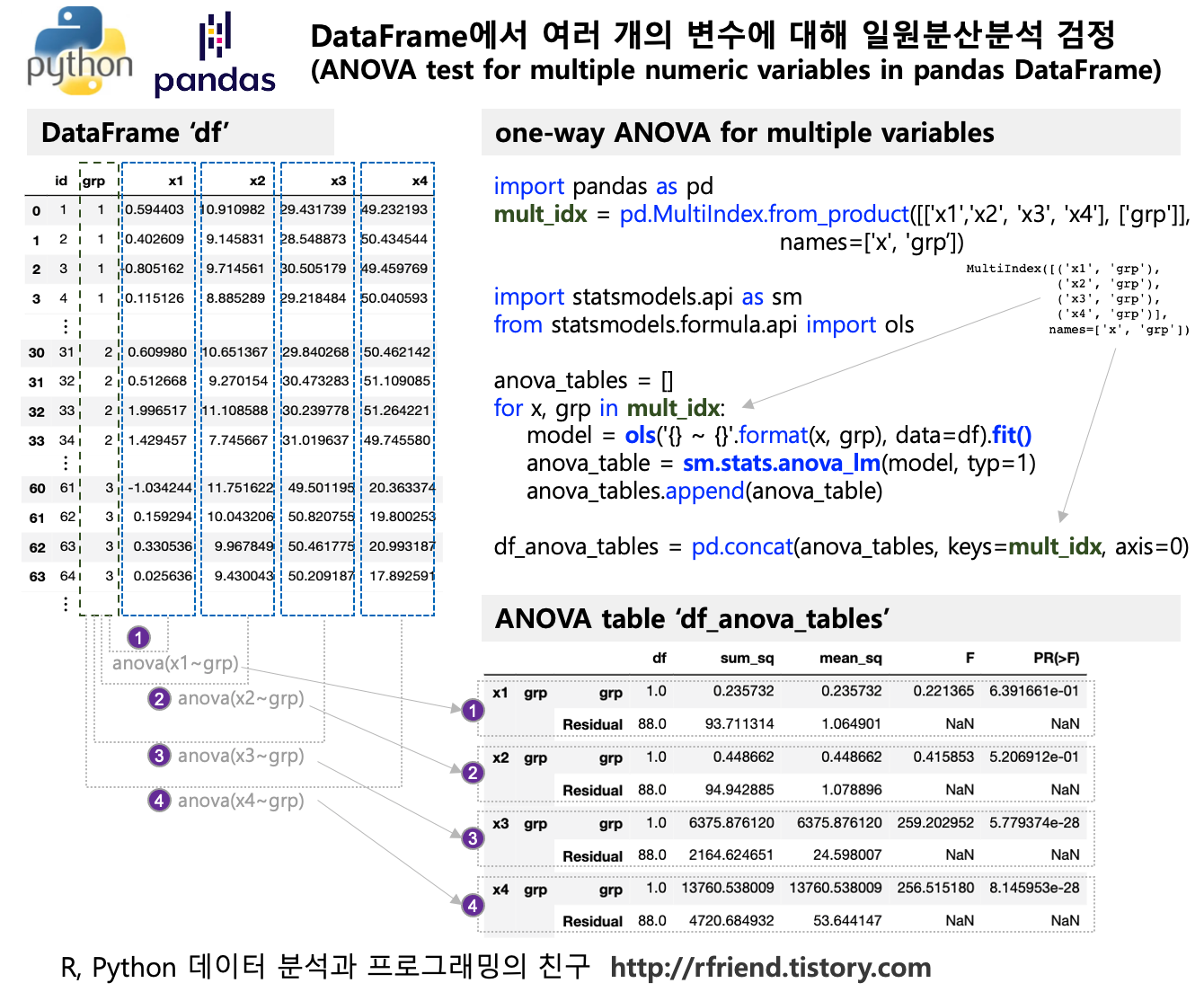
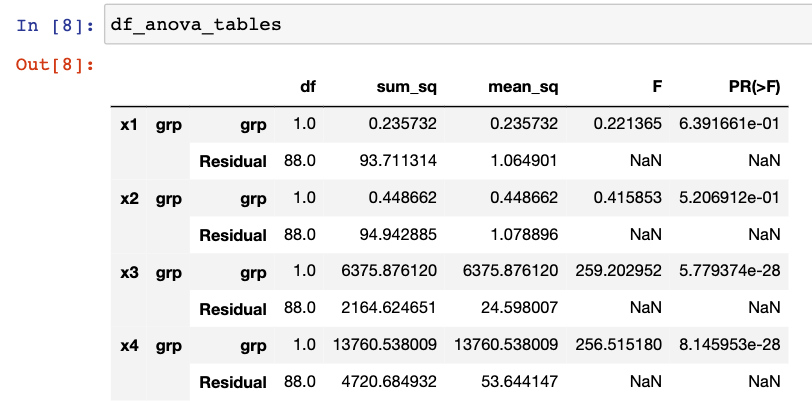
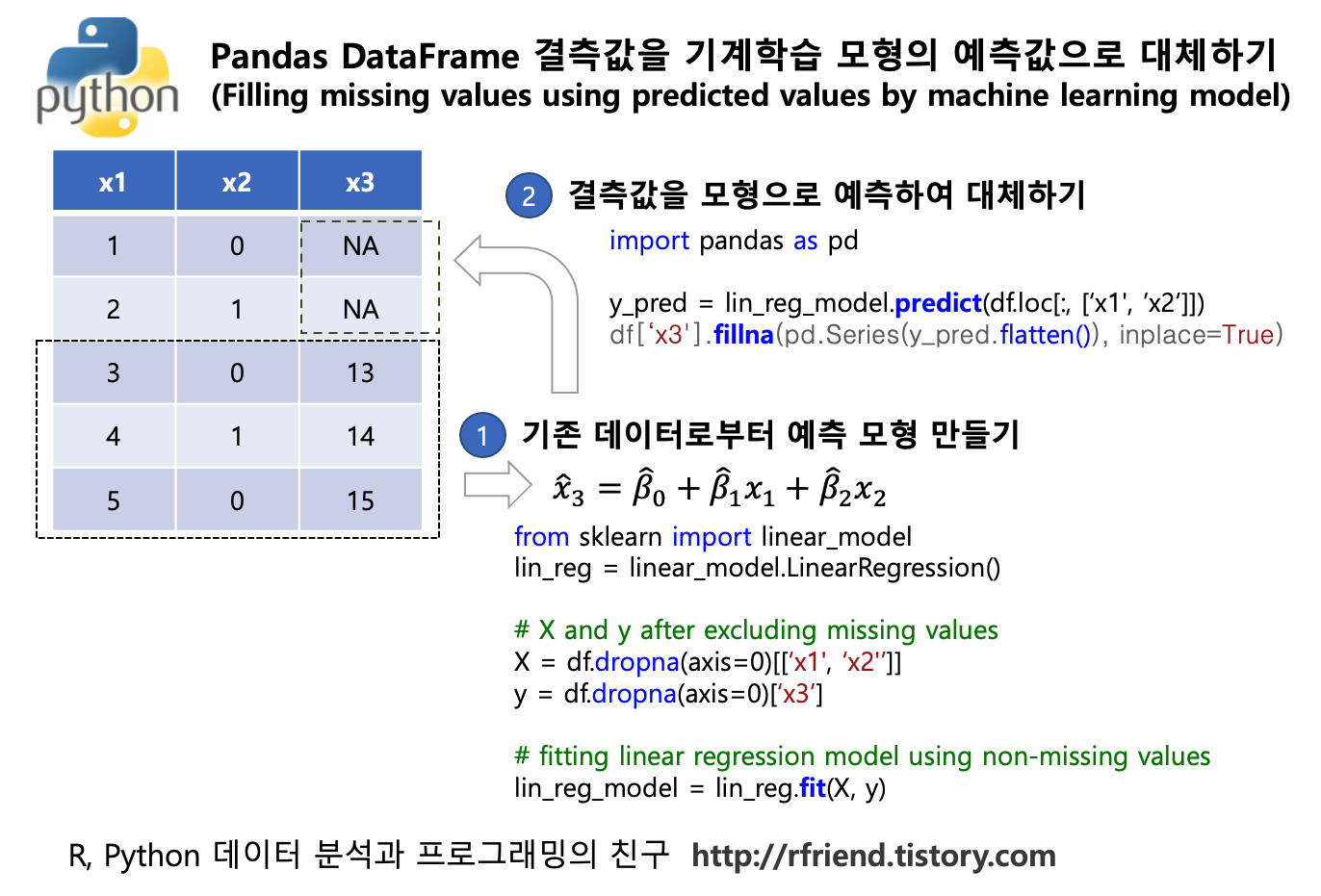
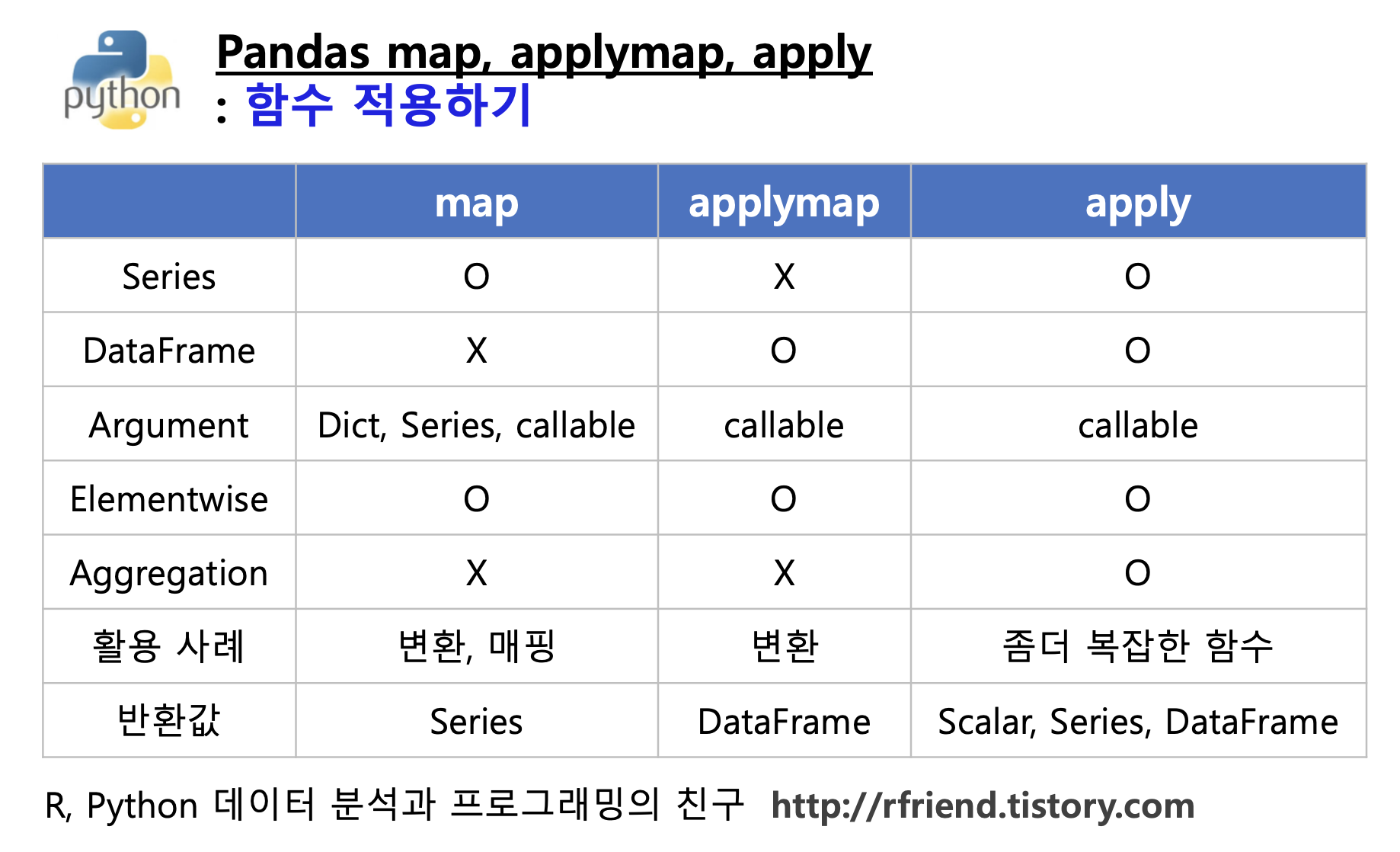
이번 포스팅에서는 Python 의 Pandas 에서 함수를 적용할 때 사용하는 map(), applymap(), apply() 메소드에 대해서 알아보겠습니다.
(1) map(): Series 에 대해 element-wise 로 함수 적용
(2) applymap(): DataFrame에 대해 element-wise 로 함수 적용
(3) apply(): DataFrame에 대해 행/열 (row/column) 로 함수 적용
(1) map(): Series 에 대해 element-wise 로 함수 적용
예제로 사용할 DataFrame을 만들어보겠습니다.
import numpy as np
import pandas as pd
## making a sample pandas DataFrame
np.random.seed(1004)
df = pd.DataFrame(
np.random.randn(4, 3),
columns=['x1', 'x2', 'x3'],
index=['A', 'B', 'C', 'D'])
print(df)
# x1 x2 x3
# A 0.594403 0.402609 -0.805162
# B 0.115126 -0.753065 -0.784118
# C 1.461576 1.576076 -0.171318
# D -0.914482 0.860139 0.358802
map() 메소드는 pandas Series 에 대해 함수를 적용할 때 사용합니다. 아래 예제는 익명 함수 lambda 로 정의한 s_formater 함수를 map() 메소드로 Series에 적용해 본 것입니다.
## pandas Series
df['x1']
# A 0.594403
# B 0.115126
# C 1.461576
# D -0.914482
# Name: x1, dtype: float64
## map: applying an element-wise function for Series
s_formater = lambda x: '%.2f'% x
df['x1'].map(s_formater)
# A 0.59
# B 0.12
# C 1.46
# D -0.91
# Name: x1, dtype: object
아래 예제에서는 Series의 인덱스를 키로 해서 매핑(mapping by index)하여 Series의 값을 변환한 것입니다.
-------------------------------------------------------
s_1 인덱스 --> s_1 값 = s_2 인덱스 --> s_2 값
-------------------------------------------------------
'a' --> 0 --> 'A'
'b' --> 1 --> 'B'
'c' --> 2 --> 'C'
-------------------------------------------------------
## 예제 pandas Series 만들기
s_1 = pd.Series({'a': 0, 'b': 1, 'c': 2})
s_2 = pd.Series({0: 'A', 1: 'B', 2: 'C'})
print(s_1)
# a 0
# b 1
# c 2
# dtype: int64
print(s_2)
# 0 A
# 1 B
# 2 C
# dtype: object
##-- 인덱스 키를 기준으로 매핑하기 (mapping by index on Series)
print(s_1.map(s_2))
# a A
# b B
# c C
# dtype: object
(2) applymap(): DataFrame에 대해 element-wise 로 함수 적용
다음 예제는 applymap() 메소드를 사용해서 익명함수 lambda 로 정의한 s_formater 함수를 DataFrame 의 각 원소에 대해 적용한 것입니다. (map() 은 Series 대상 vs. applymap()은 DataFrame 대상 element-wise 함수 적용)
## applymap: applying an element-wise for DataFrame
s_formater = lambda x: '%.2f'% x
df_2 = df.applymap(s_formater)
print(df_2)
# x1 x2 x3
# A 0.59 0.40 -0.81
# B 0.12 -0.75 -0.78
# C 1.46 1.58 -0.17
# D -0.91 0.86 0.36
(3) apply(): DataFrame에 대해 행/열 (row/column) 로 함수 적용
아래 예제는 column 기준 (axis=0), row 기준 (axis=1) 으로 익명함수 lambda로 정의한 (열 또는 행 기준, 최대값 - 최소값) 을 계산하는 함수를 apply() 메소드로 DataFrame에 대해 적용해본 것입니다.
## apply: applying a function on row/column basis of a DataFrame
f = lambda x: x.max() - x.min()
## on column basis
df.apply(f, axis=0)
# x1 2.376058
# x2 2.329141
# x3 1.163964
# dtype: float64
## on row basis
df.apply(f, axis=1)
# A 1.399565
# B 0.899243
# C 1.747393
# D 1.774621
# dtype: float64
아래 예는 apply() 메소드를 써서 DataFrame에 행(row) 기준으로 최대값을 계산하는 익명함수 lambda로 정의한 함수를 적용해서 'x_max'라는 새로운 칼럼을 추가한 것입니다.
## 새로운 칼럼 추가하기
df['x_max'] = df.apply(lambda x: x.max(), axis=1)
print(df)
# x1 x2 x3 x_max
# A 0.594403 0.402609 -0.805162 0.594403
# B 0.115126 -0.753065 -0.784118 0.115126
# C 1.461576 1.576076 -0.171318 1.576076
# D -0.914482 0.860139 0.358802 0.860139
## equivalent
df['x_max'] = df.max(axis=1)
* Pandas DataFrame에서 여러개의 칼럼에 대해 그룹을 집계를 할 때 다른 집계 함수를 적용하는 방법은 https://rfriend.tistory.com/393 를 참고하세요.
* Python의 익명 함수 lambda 에 대해서는 https://rfriend.tistory.com/366 를 참고하세요.
이번 포스팅이 많은 도움이 되었기를 바랍니다.
행복한 데이터 과학자 되세요! :-)

'Python 분석과 프로그래밍 > Python 데이터 전처리' 카테고리의 다른 글
| [Python pandas] 리스트를 행으로 변환하여 DataFrame 만들기 (0) | 2023.08.14 |
|---|---|
| [Python] 리스트와 사전 자료형을 이용해서 문자열과 숫자 매핑하기 (0) | 2023.07.16 |
| [Python] 파워포인트와 PDF 파일에서 텍스트 추출하기 (0) | 2023.03.19 |
| [Python Numpy] 반복자 enumerate() vs. 다차원 반복자np.ndenumerate() (0) | 2023.03.05 |
| [Python pandas] DataFrame.filter(): 특정 조건에 맞는 칼럼이나 행을 선택해 가져오기 (0) | 2023.01.17 |