[Python] 분산 안정화 변환과 차분으로 정상확률과정으로 변환(variance stabilization transformation and differencing for stationarity)
Python 분석과 프로그래밍/Python 통계분석 2021. 10. 31. 21:56이전 포스팅에서는
(i) 정상확률과정(stationary process)의 정의 (https://rfriend.tistory.com/691)
(ii) 통계적 가설 검증을 통한 시계열 정상성(stationarity test) 여부 확인 (https://rfriend.tistory.com/694)
하는 방법을 소개하였습니다.
ARIMA 모형과 같은 통계적 시계열 예측 모델의 경우 시계열데이터의 정상성 가정을 충족시켜야 합니다. 따라서 만약 시계열 데이터가 비정상 확률 과정 (non-stationary process) 이라면, 먼저 시계열 데이터 변환을 통해서 정상성(stationarity)을 충족시켜주어야 ARIMA 모형을 적합할 수 있습니다.
이번 포스팅에서는 Python을 사용하여
(1) 분산이 고정적이지 않은 경우 분산 안정화 변환 (variance stabilizing transformation, VST)
(2) 추세가 있는 경우 차분을 통한 추세 제거 (de-trend by differencing)
(3) 계절성이 있는 경우 계절 차분을 통한 계절성 제거 (de-seasonality by seaanl differencing)
하는 방법을 소개하겠습니다.
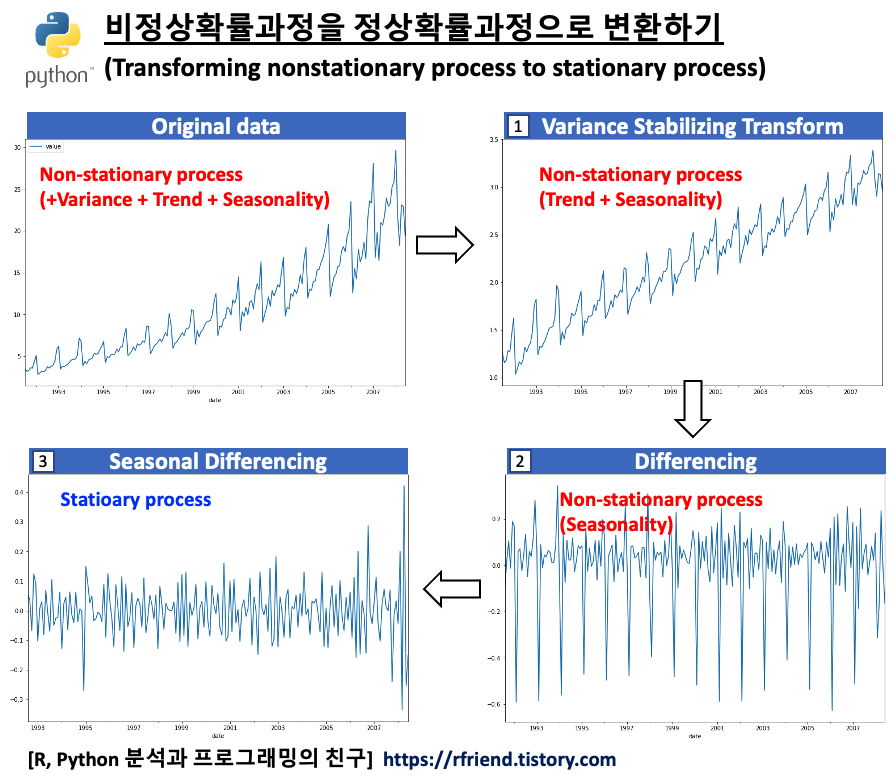
[ 비정상확률과정을 정상확률과정으로 변환하기 (Transforming non-stationary to stationary process) ]
먼저 예제로 사용할 약 판매량 (drug sales) 시계열 데이터를 가져와서 pandas DataFrame으로 만들고, 시계열 그래프를 그려보겠습니다.
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
## getting drug sales dataset
file_path = 'https://raw.githubusercontent.com/selva86/datasets/master/a10.csv'
df = pd.read_csv(file_path,
parse_dates=['date'],
index_col='date')
df.head(12)
# value
# date
# 3.526591
# 1991-08-01 3.180891
# 1991-09-01 3.252221
# 1991-10-01 3.611003
# 1991-11-01 3.565869
# 1991-12-01 4.306371
# 1992-01-01 5.088335
# 1992-02-01 2.814520
# 1992-03-01 2.985811
# 1992-04-01 3.204780
# 1992-05-01 3.127578
# 1992-06-01 3.270523
## time series plot
df.plot(figsize=[12, 8])
plt.title('Non-Stationary Process: Increasing Variance + Trend + Seasonality',
fontsize=16)
plt.show()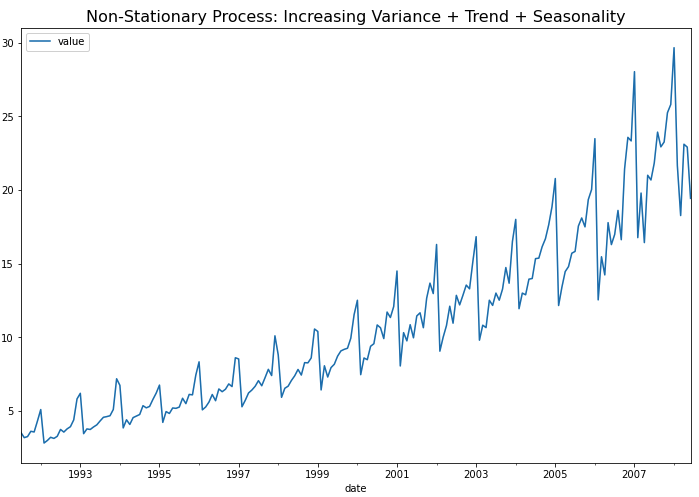
위의 시계열 그래프에서 볼 수 있는 것처럼, (a) 분산이 시간의 흐름에 따라 증가 하고 (분산이 고정이 아님), (b) 추세(trend)가 있으며, (c) 1년 주기의 계절성(seasonality)이 있으므로, 비정상확률과정(non-stationary process)입니다.
KPSS 검정을 통해서 확인해봐도 p-value가 0.01 이므로 유의수준 5% 하에서 귀무가설 (H0: 정상 시계열이다)을 기각하고, 대립가설(H1: 정상 시계열이 아니다)을 채택합니다.
## UDF for KPSS test
from statsmodels.tsa.stattools import kpss
import pandas as pd
def kpss_test(timeseries):
print("Results of KPSS Test:")
kpsstest = kpss(timeseries, regression="c", nlags="auto")
kpss_output = pd.Series(
kpsstest[0:3], index=["Test Statistic", "p-value", "Lags Used"] )
for key, value in kpsstest[3].items():
kpss_output["Critical Value (%s)" % key] = value
print(kpss_output)
## 귀무가설 (H0): 정상 시계열이다
## 대립가설 (H1): 정상 시계열이 아니다 <-- p-value 0.01
kpss_test(df)
# Results of KPSS Test:
# Test Statistic 2.013126
# p-value 0.010000
# Lags Used 9.000000
# Critical Value (10%) 0.347000
# Critical Value (5%) 0.463000
# Critical Value (2.5%) 0.574000
# Critical Value (1%) 0.739000
# dtype: float64
(1) 분산이 고정적이지 않은 경우 분산 안정화 변환 (variance stabilizing transformation, VST)
분산이 고정적이지 않은 경우 멱 변환(Power Transformation)을 통해서 분산을 안정화(variance stabilization) 시켜줍니다. 분산이 고정적이지 않고 추세가 있는 경우 분산 안정화를 추세 제거보다 먼저 해줍니다. 왜냐하면 추세를 제거하기 위해 차분(differencing)을 해줄 때 음수(-)가 생길 수 있기 때문입니다.
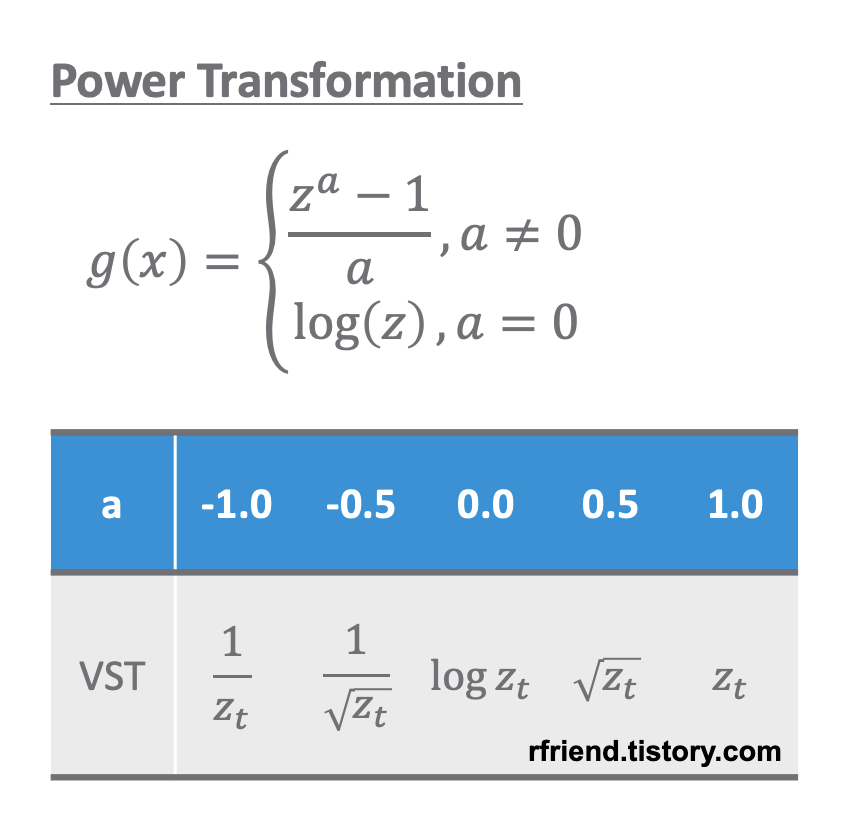
원래의 시계열 데이터의 분산 형태에 따라서 적합한 멱 변환(power transformation)을 선택해서 정상확률과정으로 변환해줄 수 있습니다. 아래의 예제 시도표를 참고하세요.
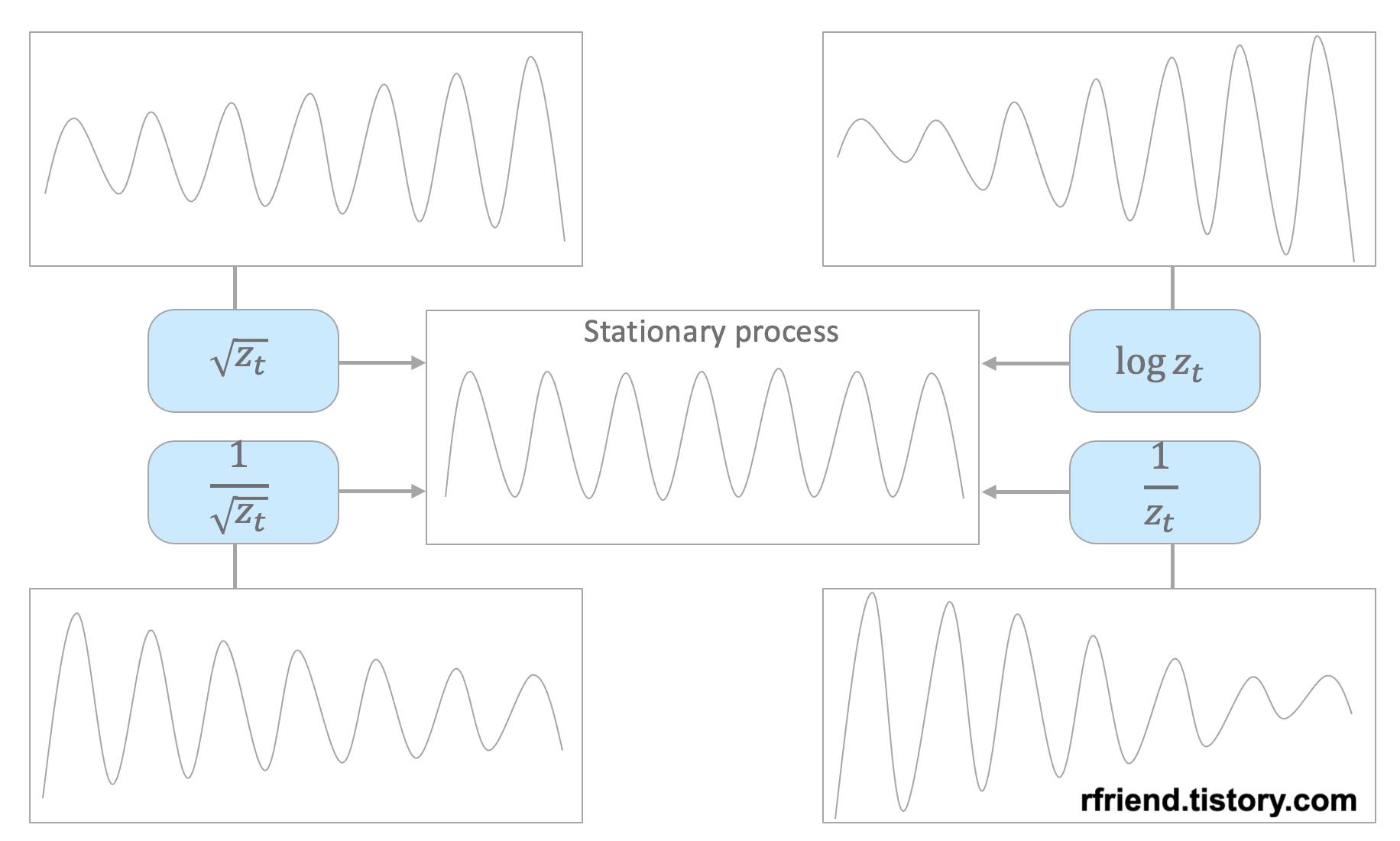
이번 포스팅에서 사용하는 예제는 시간이 흐릴수록 분산이 점점 커지는 형태를 띠고 있으므로 로그 변환(log transformation) 이나 제곱근 변환 (root transformation) 을 해주면 정상 시계열로 변환이 되겠네요. 아래 코드에서는 자연로그를 취해서 로그 변환을 해주었습니다.
## Variance Stabilizing Transformation (VST) by Taking Logarithm
df_vst = np.log(df.value)
df_vst.head()
# date
# 1991-07-01 1.260332
# 1991-08-01 1.157161
# 1991-09-01 1.179338
# 1991-10-01 1.283986
# 1991-11-01 1.271408
# Name: value, dtype: float64
## plotting
df_vst.plot(figsize=(12, 8))
plt.title("Variance Stabilizing Transformation by taking Logarithm",
fontsize=16)
plt.show()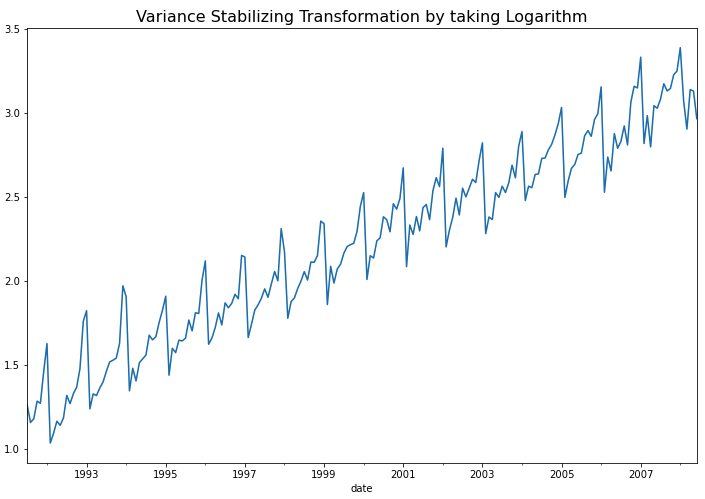
위의 시도표를 보면 시간이 경과해도 분산이 안정화되었음을 알 수 있습니다. KPSS 검정을 한번 더 해주면 아직 추세(trend)와 계절성(seasonality)가 남아있으므로 여전히 비정상확률과정을 따른다고 나옵니다.
## 귀무가설 (H0): 정상 시계열이다
## 대립가설 (H1): 정상 시계열이 아니다 <-- p-value 0.01
kpss_test(df_vst)
# Results of KPSS Test:
# Test Statistic 2.118189
# p-value 0.010000
# Lags Used 9.000000
# Critical Value (10%) 0.347000
# Critical Value (5%) 0.463000
# Critical Value (2.5%) 0.574000
# Critical Value (1%) 0.739000
# dtype: float64
(2) 추세가 있는 경우 차분을 통한 추세 제거 (de-trend by differencing)
차분(differencing)은 현재의 시계열 값에서 시차 t 만큼의 이전 값을 빼주는 것입니다.
1차 차분 = Delta1_Z(t) = Z(t) - Z(t-1)
2차 차분 = Delta2_Z(t) = Z(t) - Z(t-1) - (Z(t-1) - Z(t-2)) = Z(t) - 2Z(t-1) + Z(t-2)
Python의 diff() 메소드를 사용해서 차분을 해줄 수 있습니다. 이때 차분의 차수 만큼 결측값이 생기는 데요, dropna() 메소드를 사용해서 결측값은 제거해주었습니다.
## De-trend by Differencing
df_vst_diff1 = df_vst.diff(1).dropna()
df_vst_diff1.plot(figsize=(12, 8))
plt.title("De-trend by 1st order Differencing", fontsize=16)
plt.show()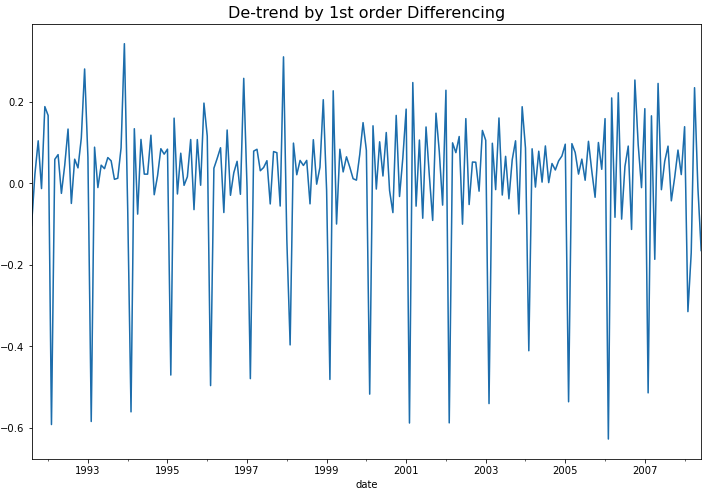
위의 시도표를 보면 1차 차분(1st order differencing)을 통해서 이제 추세(trend)도 제거되었음을 알 수 있습니다. 하지만 아직 계절성(seasonality)이 남아있어서 정상성 조건은 만족하지 않겠네요. 그런데 아래에 KPSS 검정을 해보니 p-value가 0.10 으로서 유의수준 5% 하에서 정상성을 만족한다고 나왔네요. ^^;
## 귀무가설 (H0): 정상 시계열이다 <-- p-value 0.10
## 대립가설 (H1): 정상 시계열이 아니다
kpss_test(df_vst_diff1)
# Results of KPSS Test:
# Test Statistic 0.121364
# p-value 0.100000
# Lags Used 37.000000
# Critical Value (10%) 0.347000
# Critical Value (5%) 0.463000
# Critical Value (2.5%) 0.574000
# Critical Value (1%) 0.739000
# dtype: float64
(3) 계절성이 있는 경우 계절 차분을 통한 계절성 제거 (de-seasonality by seaanl differencing)
아직 남아있는 계절성(seasonality)을 계절 차분(seasonal differencing)을 사용해서 제거해보겠습니다. 1년 12개월 주기의 계절성을 띠고 있으므로 diff(12) 함수로 계절 차분을 실시하고, 12개의 결측값이 생기는데요 dropna() 로 결측값은 제거해주었습니다.
## Stationary Process: De-seasonality by Seasonal Differencing
df_vst_diff1_diff12 = df_vst_diff1.diff(12).dropna()
## plotting
df_vst_diff1_diff12.plot(figsize=(12, 8))
plt.title("De-seasonality by Seasonal Differencing",
fontsize=16)
plt.show()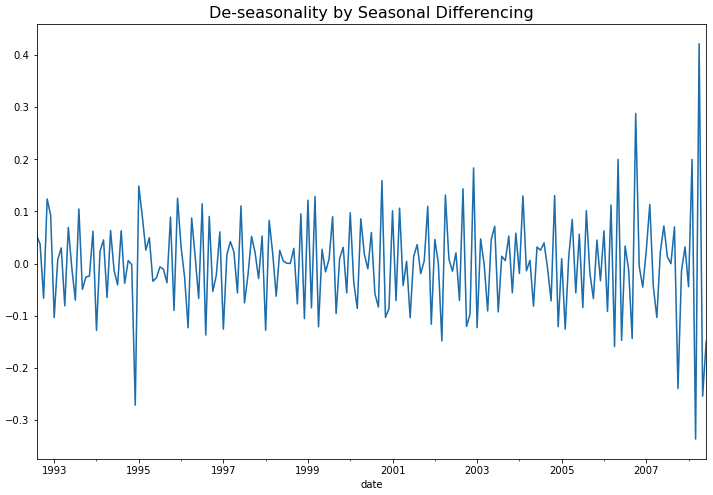
위의 시도표를 보면 이제 계절성도 제거가 되어서 정상 시계열처럼 보이네요. 아래에 KPSS 검정을 해보니 p-value 가 0.10 으로서, 유의수준 5% 하에서 귀무가설(H0: 정상 시계열이다)을 채택할 수 있겠네요.
## 귀무가설 (H0): 정상 시계열이다 <-- p-value 0.10
## 대립가설 (H1): 정상 시계열이 아니다
kpss_test(df_vst_diff1_diff12)
# Results of KPSS Test:
# Test Statistic 0.08535
# p-value 0.10000
# Lags Used 8.00000
# Critical Value (10%) 0.34700
# Critical Value (5%) 0.46300
# Critical Value (2.5%) 0.57400
# Critical Value (1%) 0.73900
# dtype: float64
이제 비정상 시계열(non-stationary process)이었던 원래 데이터를 (1) log transformation을 통한 분산 안정화, (2) 차분(differencing)을 통한 추세 제거, (3) 계절 차분(seasonal differencing)을 통한 계절성 제거를 모두 마쳐서 정상 시계열(stationary process) 로 변환을 마쳤으므로, ARIMA 통계 모형을 적합할 수 있게 되었습니다.
이번 포스팅이 많은 도움이 되었기를 바랍니다.
행복한 데이터 과학자 되세요! :-)






