[R data.table] 조건이 있는 상태에서 Key를 기준으로 데이터셋 합치기 (Conditional Joins)
R 분석과 프로그래밍/R 데이터 전처리 2021. 1. 31. 23:36지난 포스팅에서는 R data.table 패키지에서 선형회귀 모델의 오른쪽 부분의 변수 조합을 .SD, .SDcols, lapply(), sapply()를 사용해서 간단하게 단 몇줄의 코드로 처리하는 방법(rfriend.tistory.com/609)을 소개하였습니다.
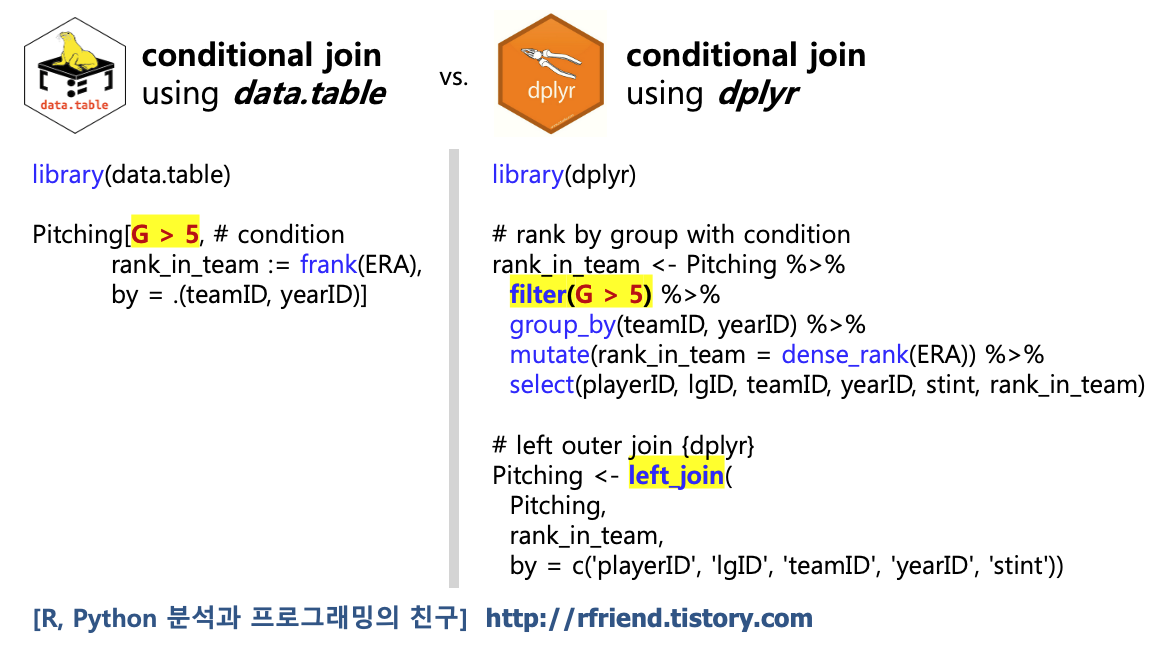
이번 포스팅에서는 R data.table 패키지에서 '조건이 있는 상태에서 Key를 기준으로 데이터셋을 Left Join 하는 방법 (Conditional Joins)'을 소개하겠습니다. 그리고 base R이나 dplyr 대비 data.table의 조건이 있는 경우의 데이터셋끼리 병합이 얼마나 간단한지 비교를 해보겠습니다. 이번 포스팅은 R data.table의 Vignettes 을 참조하였습니다.
(1) data.table을 이용한 조건이 있는 경우의 Left Join
(2) dplyr을 이용한 조건이 있는 경우의 Left Join 비교
먼저, data.table 패키지를 불러오고, 예제로 사용할 데이터로 Lahman 패키지에 들어있는 야구 투구 통계 데이터인 'Pitching' 데이터셋과 야구 팀 성적 통계 데이터인 'Teams' 데이터셋을 참조해서 data.table과 data.frame으로 만들어보겠습니다.
library(data.table)
## Lahman database on baseball
#install.packages("Lahman")
library(Lahman)
data("Pitching")
## data.frame
Pitching_df <- data.frame(Pitching)
## data.table
setDT(Pitching)
str(Pitching)
# Classes 'data.table' and 'data.frame': 47628 obs. of 30 variables:
# $ playerID: chr "bechtge01" "brainas01" "fergubo01" "fishech01" ...
# $ yearID : int 1871 1871 1871 1871 1871 1871 1871 1871 1871 1871 ...
# $ stint : int 1 1 1 1 1 1 1 1 1 1 ...
# $ teamID : Factor w/ 149 levels "ALT","ANA","ARI",..: 97 142 90 111 90 136 111 56 97 136 ...
# $ lgID : Factor w/ 7 levels "AA","AL","FL",..: 4 4 4 4 4 4 4 4 4 4 ...
# $ W : int 1 12 0 4 0 0 0 6 18 12 ...
# $ L : int 2 15 0 16 1 0 1 11 5 15 ...
# $ G : int 3 30 1 24 1 1 3 19 25 29 ...
# $ GS : int 3 30 0 24 1 0 1 19 25 29 ...
# $ CG : int 2 30 0 22 1 0 1 19 25 28 ...
# $ SHO : int 0 0 0 1 0 0 0 1 0 0 ...
# $ SV : int 0 0 0 0 0 0 0 0 0 0 ...
# $ IPouts : int 78 792 3 639 27 3 39 507 666 747 ...
# $ H : int 43 361 8 295 20 1 20 261 285 430 ...
# $ ER : int 23 132 3 103 10 0 5 97 113 153 ...
# $ HR : int 0 4 0 3 0 0 0 5 3 4 ...
# $ BB : int 11 37 0 31 3 0 3 21 40 75 ...
# $ SO : int 1 13 0 15 0 0 1 17 15 12 ...
# $ BAOpp : num NA NA NA NA NA NA NA NA NA NA ...
# $ ERA : num 7.96 4.5 27 4.35 10 0 3.46 5.17 4.58 5.53 ...
# $ IBB : int NA NA NA NA NA NA NA NA NA NA ...
# $ WP : int 7 7 2 20 0 0 1 15 3 44 ...
# $ HBP : int NA NA NA NA NA NA NA NA NA NA ...
# $ BK : int 0 0 0 0 0 0 0 2 0 0 ...
# $ BFP : int 146 1291 14 1080 57 3 70 876 1059 1334 ...
# $ GF : int 0 0 0 1 0 1 1 0 0 0 ...
# $ R : int 42 292 9 257 21 0 30 243 223 362 ...
# $ SH : int NA NA NA NA NA NA NA NA NA NA ...
# $ SF : int NA NA NA NA NA NA NA NA NA NA ...
# $ GIDP : int NA NA NA NA NA NA NA NA NA NA ...
# - attr(*, ".internal.selfref")=<externalptr>
data("Teams")
setDT(Teams)
str(Teams)
# Classes 'data.table' and 'data.frame': 2925 obs. of 48 variables:
# $ yearID : int 1871 1871 1871 1871 1871 1871 1871 1871 1871 1872 ...
# $ lgID : Factor w/ 7 levels "AA","AL","FL",..: 4 4 4 4 4 4 4 4 4 4 ...
# $ teamID : Factor w/ 149 levels "ALT","ANA","ARI",..: 24 31 39 56 90 97 111 136 142 8 ...
# $ franchID : Factor w/ 120 levels "ALT","ANA","ARI",..: 13 36 25 56 70 85 91 109 77 9 ...
# $ divID : chr NA NA NA NA ...
# $ Rank : int 3 2 8 7 5 1 9 6 4 2 ...
# $ G : int 31 28 29 19 33 28 25 29 32 58 ...
# $ Ghome : int NA NA NA NA NA NA NA NA NA NA ...
# $ W : int 20 19 10 7 16 21 4 13 15 35 ...
# $ L : int 10 9 19 12 17 7 21 15 15 19 ...
# $ DivWin : chr NA NA NA NA ...
# $ WCWin : chr NA NA NA NA ...
# $ LgWin : chr "N" "N" "N" "N" ...
# $ WSWin : chr NA NA NA NA ...
# $ R : int 401 302 249 137 302 376 231 351 310 617 ...
# $ AB : int 1372 1196 1186 746 1404 1281 1036 1248 1353 2571 ...
# $ H : int 426 323 328 178 403 410 274 384 375 753 ...
# $ X2B : int 70 52 35 19 43 66 44 51 54 106 ...
# $ X3B : int 37 21 40 8 21 27 25 34 26 31 ...
# $ HR : int 3 10 7 2 1 9 3 6 6 14 ...
# $ BB : int 60 60 26 33 33 46 38 49 48 29 ...
# $ SO : int 19 22 25 9 15 23 30 19 13 28 ...
# $ SB : int 73 69 18 16 46 56 53 62 48 53 ...
# $ CS : int 16 21 8 4 15 12 10 24 13 18 ...
# $ HBP : int NA NA NA NA NA NA NA NA NA NA ...
# $ SF : int NA NA NA NA NA NA NA NA NA NA ...
# $ RA : int 303 241 341 243 313 266 287 362 303 434 ...
# $ ER : int 109 77 116 97 121 137 108 153 137 166 ...
# $ ERA : num 3.55 2.76 4.11 5.17 3.72 4.95 4.3 5.51 4.37 2.9 ...
# $ CG : int 22 25 23 19 32 27 23 28 32 48 ...
# $ SHO : int 1 0 0 1 1 0 1 0 0 1 ...
# $ SV : int 3 1 0 0 0 0 0 0 0 1 ...
# $ IPouts : int 828 753 762 507 879 747 678 750 846 1548 ...
# $ HA : int 367 308 346 261 373 329 315 431 371 573 ...
# $ HRA : int 2 6 13 5 7 3 3 4 4 3 ...
# $ BBA : int 42 28 53 21 42 53 34 75 45 63 ...
# $ SOA : int 23 22 34 17 22 16 16 12 13 77 ...
# $ E : int 243 229 234 163 235 194 220 198 218 432 ...
# $ DP : int 24 16 15 8 14 13 14 22 20 22 ...
# $ FP : num 0.834 0.829 0.818 0.803 0.84 0.845 0.821 0.845 0.85 0.83 ...
# $ name : chr "Boston Red Stockings" "Chicago White Stockings" "Cleveland Forest Citys" "Fort Wayne Kekiongas" ...
# $ park : chr "South End Grounds I" "Union Base-Ball Grounds" "National Association Grounds" "Hamilton Field" ...
# $ attendance : int NA NA NA NA NA NA NA NA NA NA ...
# $ BPF : int 103 104 96 101 90 102 97 101 94 106 ...
# $ PPF : int 98 102 100 107 88 98 99 100 98 102 ...
# $ teamIDBR : chr "BOS" "CHI" "CLE" "KEK" ...
# $ teamIDlahman45: chr "BS1" "CH1" "CL1" "FW1" ...
# $ teamIDretro : chr "BS1" "CH1" "CL1" "FW1" ...
# - attr(*, ".internal.selfref")=<externalptr>
(1) data.table을 이용한 조건이 있는 경우의 Left Join
(1-1) 참여 경기 수 조건 하에 팀별 연도별 평균자책점 순위 (rank_in_team)
(조건) G > 5 : 6개 이상의 경기(G, game)에 참여하여 공을 던진 투수에 한하여,
(연산) ERA (평균 자책점, Earned Run Average)의 순위를 구해서 rank_in_team 변수를 만들되,
(그룹 기준) 'teamID' & 'yearID' 그룹 별로 ERA 순위(frank(ERA))를 구하여라.
조건, 연산, 그룹 기준이 모두 Pitching[조건, 연산, 그룹 기준] 의 구문으로 해서 단 한줄로 모두 처리가 가능합니다. 간결함의 끝판왕이라고나 할까요!
## -- rank in team using frank() function by teamID & yearID groups
## to exclude pitchers with exceptional performance in a few games,
## subset first; then define rank of pitchers within their team each year
## (in general, we should put more care into the 'ties.method' of frank)
Pitching[G > 5, rank_in_team := frank(ERA), by = .(teamID, yearID)]
head(Pitching)
# playerID yearID stint teamID lgID W L G GS CG SHO SV IPouts H ER HR BB SO BAOpp ERA
# 1: bechtge01 1871 1 PH1 NA 1 2 3 3 2 0 0 78 43 23 0 11 1 NA 7.96
# 2: brainas01 1871 1 WS3 NA 12 15 30 30 30 0 0 792 361 132 4 37 13 NA 4.50
# 3: fergubo01 1871 1 NY2 NA 0 0 1 0 0 0 0 3 8 3 0 0 0 NA 27.00
# 4: fishech01 1871 1 RC1 NA 4 16 24 24 22 1 0 639 295 103 3 31 15 NA 4.35
# 5: fleetfr01 1871 1 NY2 NA 0 1 1 1 1 0 0 27 20 10 0 3 0 NA 10.00
# 6: flowedi01 1871 1 TRO NA 0 0 1 0 0 0 0 3 1 0 0 0 0 NA 0.00
# IBB WP HBP BK BFP GF R SH SF GIDP rank_in_team
# 1: NA 7 NA 0 146 0 42 NA NA NA NA
# 2: NA 7 NA 0 1291 0 292 NA NA NA 1
# 3: NA 2 NA 0 14 0 9 NA NA NA NA
# 4: NA 20 NA 0 1080 1 257 NA NA NA 1
# 5: NA 0 NA 0 57 0 21 NA NA NA NA
# 6: NA 0 NA 0 3 1 0 NA NA NA NA
(1-2) 팀내 순위 조건하에 다른 데이터셋에 앴는 팀 성적 (team_performance) Left join
(조건) Pitching 데이터셋에서 팀/연도별로 순위가 1등인 투수에 한해 (rank_in_team == 1)
(연산) Teams 데이터셋의 순위(Rank)를 가져다가 Pitching 데이터셋에 team_performance 이름의 변수로 만들되,
(병합 기준) Pitching 과 Teams 데이터셋의 'teamID'와 'yearID'를 기준으로 매칭하시오.
Pitching[rank_in_team == 1, # condition for rows
team_performance := Teams[.SD, Rank,
on = c('teamID', 'yearID')] # left join by keys
]
head(Pitching)
# playerID yearID stint teamID lgID W L G GS CG SHO SV IPouts H ER HR BB SO BAOpp ERA
# 1: bechtge01 1871 1 PH1 NA 1 2 3 3 2 0 0 78 43 23 0 11 1 NA 7.96
# 2: brainas01 1871 1 WS3 NA 12 15 30 30 30 0 0 792 361 132 4 37 13 NA 4.50
# 3: fergubo01 1871 1 NY2 NA 0 0 1 0 0 0 0 3 8 3 0 0 0 NA 27.00
# 4: fishech01 1871 1 RC1 NA 4 16 24 24 22 1 0 639 295 103 3 31 15 NA 4.35
# 5: fleetfr01 1871 1 NY2 NA 0 1 1 1 1 0 0 27 20 10 0 3 0 NA 10.00
# 6: flowedi01 1871 1 TRO NA 0 0 1 0 0 0 0 3 1 0 0 0 0 NA 0.00
# IBB WP HBP BK BFP GF R SH SF GIDP rank_in_team team_performance
# 1: NA 7 NA 0 146 0 42 NA NA NA NA NA
# 2: NA 7 NA 0 1291 0 292 NA NA NA 1 4
# 3: NA 2 NA 0 14 0 9 NA NA NA NA NA
# 4: NA 20 NA 0 1080 1 257 NA NA NA 1 9
# 5: NA 0 NA 0 57 0 21 NA NA NA NA NA
# 6: NA 0 NA 0 3 1 0 NA NA NA NA NA
(2) dplyr을 이용한 조건이 있는 경우의 Left Join 비교
(2-1) 참여 경기 수 조건하에 팀별 연도별 평균자책점 순위 (rank_in_team)
이제 위의 (1-1)에서 data.table로 수행했던 것과 동일한 과업을 dplyr 패키지로 수행해보겠습니다.
(조건) G > 5 : 6개 이상의 경기(G, game)에 참여하여 공을 던진 투수에 한하여,
(연산) ERA (평균 자책점, Earned Run Average)의 순위를 구해서 rank_in_team 변수를 만들되,
(그룹 기준) 'teamID' & 'yearID' 그룹 별로 ERA 순위(frank(ERA))를 구하여라.
1단계에서 G > 5 라는 조건으로 filter() 를 하여 그룹별로 rank_in_team 을 구해서 별도의 'rank_in_team_df' data.frame을 만든 후에, --> 2단계에서 이를 원본 'Pitching_df'에 left_join() 을 해서 left join 병합을 해주었습니다. G > 5 라는 조건(condition)이 들어감으로써 2단계로 나누어서 진행이 되다보니 코드가 길어졌습니다.
## -- doing the same operation using 'dplyr'
library(dplyr)
## -- rank in team by teamID & yearID group using dense_rank() window function
rank_in_team_df <- Pitching_df %>%
filter(G > 5) %>%
group_by(teamID, yearID) %>%
mutate(rank_in_team = dense_rank(ERA)) %>%
select(playerID, lgID, teamID, yearID, stint, rank_in_team)
## left outer join {dplyr}
Pitching_df <- left_join(Pitching_df,
rank_in_team_df,
by = c('playerID', 'lgID', 'teamID', 'yearID', 'stint'))
# ## left outer join {base}
# Pitching_df <- merge(x = Pitching_df,
# y = rank_in_team_df,
# by = c('playerID', 'lgID', 'teamID', 'yearID', 'stint'),
# all.x = TRUE, all.y = FALSE)
head(Pitching_df)
# playerID yearID stint teamID lgID W L G GS CG SHO SV IPouts H ER HR BB SO BAOpp ERA IBB
# 1 bechtge01 1871 1 PH1 NA 1 2 3 3 2 0 0 78 43 23 0 11 1 NA 7.96 NA
# 2 brainas01 1871 1 WS3 NA 12 15 30 30 30 0 0 792 361 132 4 37 13 NA 4.50 NA
# 3 fergubo01 1871 1 NY2 NA 0 0 1 0 0 0 0 3 8 3 0 0 0 NA 27.00 NA
# 4 fishech01 1871 1 RC1 NA 4 16 24 24 22 1 0 639 295 103 3 31 15 NA 4.35 NA
# 5 fleetfr01 1871 1 NY2 NA 0 1 1 1 1 0 0 27 20 10 0 3 0 NA 10.00 NA
# 6 flowedi01 1871 1 TRO NA 0 0 1 0 0 0 0 3 1 0 0 0 0 NA 0.00 NA
# WP HBP BK BFP GF R SH SF GIDP rank_in_team
# 1 7 NA 0 146 0 42 NA NA NA NA
# 2 7 NA 0 1291 0 292 NA NA NA 1
# 3 2 NA 0 14 0 9 NA NA NA NA
# 4 20 NA 0 1080 1 257 NA NA NA 1
# 5 0 NA 0 57 0 21 NA NA NA NA
# 6 0 NA 0 3 1 0 NA NA NA NA
(2-2) 팀내 순위 조건하에 다른 데이터셋에 앴는 팀 성적 (team_performance) Left join
이제 위의 (1-2)에서 data.table로 했던 것과 똑같은 과업을 dplyr로 해보겠습니다.
(조건) Pitching 데이터셋에서 팀/연도별로 순위가 1등인 투수에 한해 (rank_in_team == 1) (연산) Teams 데이터셋의 순위(Rank)를 가져다가 Pitching 데이터셋에 team_performance 이름의 변수로 만들되,
(병합 기준) Pitching 과 Teams 데이터셋의 'teamID'와 'yearID'를 기준으로 매칭하시오.
아래처럼 dplyr로 하게 되면 '팀/연도별로 순위가 1등인 투수에 한해(rank_in_team==1)' 라는 조건을 충족시키기 위해서 조건절을 포함한 ifelse() 절을 한번 더 수행해줘야 합니다.
## -- merging team performance
Pitching_df <- left_join(Pitching_df,
Teams[, c('teamID', 'yearID', 'Rank')],
by = c('teamID', 'yearID'))
## condition: rank_in_team == 1
Pitching_df$team_performance <- ifelse(Pitching_df$rank_in_team == 1,
Pitching_df$Rank, # if TRUE
NA) # if FALSE
head(Pitching_df)
# bechtge01 1871 1 PH1 NA 1 2 3 3 2 0 0 78 43 23 0 11 1 NA 7.96 NA
# 2 brainas01 1871 1 WS3 NA 12 15 30 30 30 0 0 792 361 132 4 37 13 NA 4.50 NA
# 3 fergubo01 1871 1 NY2 NA 0 0 1 0 0 0 0 3 8 3 0 0 0 NA 27.00 NA
# 4 fishech01 1871 1 RC1 NA 4 16 24 24 22 1 0 639 295 103 3 31 15 NA 4.35 NA
# 5 fleetfr01 1871 1 NY2 NA 0 1 1 1 1 0 0 27 20 10 0 3 0 NA 10.00 NA
# 6 flowedi01 1871 1 TRO NA 0 0 1 0 0 0 0 3 1 0 0 0 0 NA 0.00 NA
# WP HBP BK BFP GF R SH SF GIDP rank_in_team Rank team_performance
# 1 7 NA 0 146 0 42 NA NA NA NA 1 NA
# 2 7 NA 0 1291 0 292 NA NA NA 1 4 4
# 3 2 NA 0 14 0 9 NA NA NA NA 5 NA
# 4 20 NA 0 1080 1 257 NA NA NA 1 9 9
# 5 0 NA 0 57 0 21 NA NA NA NA 5 NA
# 6 0 NA 0 3 1 0 NA NA NA NA 6 NA
위에서 소개했던 똑같은 과업을 수행하는 data.table과 dplyr의 조건있는 데이터셋 병합(conditional left join) 예제 코드를 나란히 제시해서 비교해보면 아래와 같습니다. data.table이 dplyr (혹은 base R) 대비 조건있는 데이터셋 병합의 경우 비교할 수 없을 정도로 훨~씬 코드가 간결합니다!
|
구분 |
data.table |
dplyr |
|
(1) conditional left join by key w/ a list |
library(data.table) rank_in_team := frank(ERA), by = .(teamID, yearID)] |
library(dplyr) filter(G > 5) %>% group_by(teamID, yearID) %>% mutate(rank_in_team = dense_rank(ERA)) %>% select(playerID, lgID, teamID, yearID, stint, rank_in_team) ## left outer join {dplyr} Pitching_df <- left_join( Pitching_df, rank_in_team_df, by = c('playerID', 'lgID', 'teamID', 'yearID', 'stint')) |
|
(2) conditional left join by key w/ a data.table |
Pitching[rank_in_team == 1, team_performance := Teams[ .SD, Rank, |
## -- merging team performance Pitching_df <- left_join( Pitching_df, Teams[, c('teamID', 'yearID', 'Rank')], by = c('teamID', 'yearID')) ## condition: rank_in_team == 1 Pitching_df$team_performance <- ifelse( Pitching_df$rank_in_team == 1, Pitching_df$Rank, # if TRUE NA) # if FALSE |
[ Reference ]
* R data.table vignettes 'Using .SD for Data Analysis'
: cran.r-project.org/web/packages/data.table/vignettes/datatable-sd-usage.html
이번 포스팅이 많은 도움이 되었기를 바랍니다.
행복한 데이터 과학자 되세요! :-)

'R 분석과 프로그래밍 > R 데이터 전처리' 카테고리의 다른 글
| [R data.table] 그룹별 최소값 행, 최대값 행 가져오기 (Group Optima) (0) | 2021.01.31 |
|---|---|
| [R data.table] .SD[], by를 사용해 그룹별로 부분집합 가져오기 (Group Subsetting) (0) | 2021.01.31 |
| [R data.table] 선형회귀 모델의 오른쪽 부분(model's right-hand side)의 변수 조합을 간단하게 다루기 (4) | 2021.01.31 |
| [R data.table] .SDcols 로 일부 칼럼 가져오기 (Column subsetting using .SDcols) (6) | 2021.01.31 |
| [R data.table] 그룹별 관측치 개수 별로 DataTable을 구분해서 생성하기 (0) | 2021.01.30 |