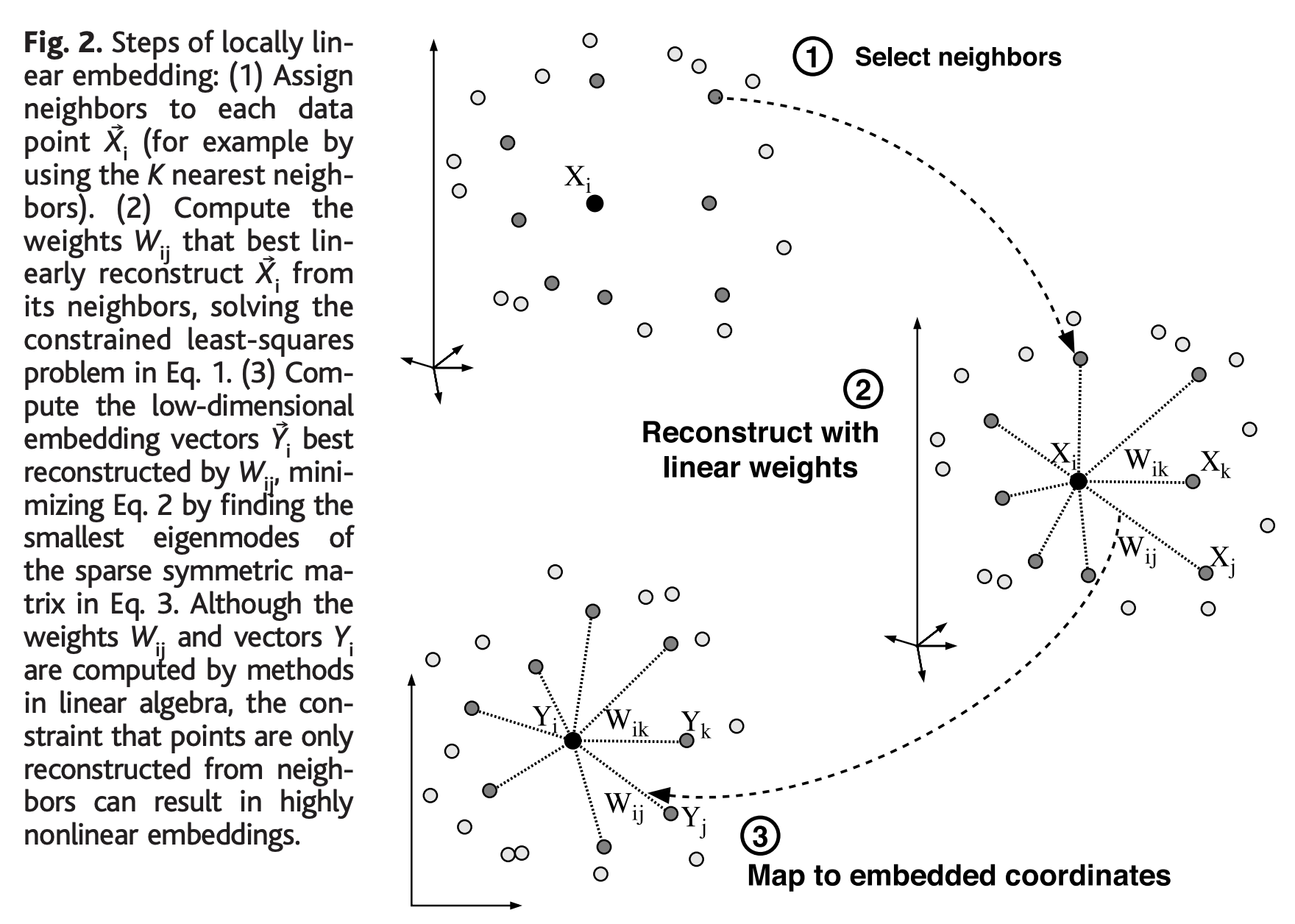
[Python] Regularization, LASSO, Ridge, Elastic Net 소개 및 Python 실습
Python 분석과 프로그래밍/Python 기계학습 2023. 12. 11. 19:14기계학습에서 Regularization 은 Penalization, Constraing method, Shrinkage methods 라고도 하는데요, "규제하다", "벌점을 주다", "제약을 가하다", "축소시키다" 등의 억압을 가하는 의미를 가지고 있습니다.
보통 Regularization을 "정규화"라고 번역하는 경우가 많던데요, Normalization 도 "정규화"라고 번역을 하곤 해서 개념이 서로 혼동되기 쉬운 면이 있어서요, 이번 포스팅에서는 그냥 Regularization 을 그대로 사용하겠습니다. 굳이 번역하라면 "규제화"나 "벌점화", "(가중치) 축소화" 등으로 번역할 수 있을 거 같습니다.
이번 포스팅에서는 Regularization 이 무엇이며 왜 사용하는지에 대해 알아보겠습니다. 또한 Regularization 기술 및 그 사용 방법에 대해 소개할 것입니다.
1. 기계 학습에서의 Regularization 은 무엇이고 왜 사용하는가?
2. L1 Regularization (LASSO Regression)
3. L2 Regularization (Ridge Regression
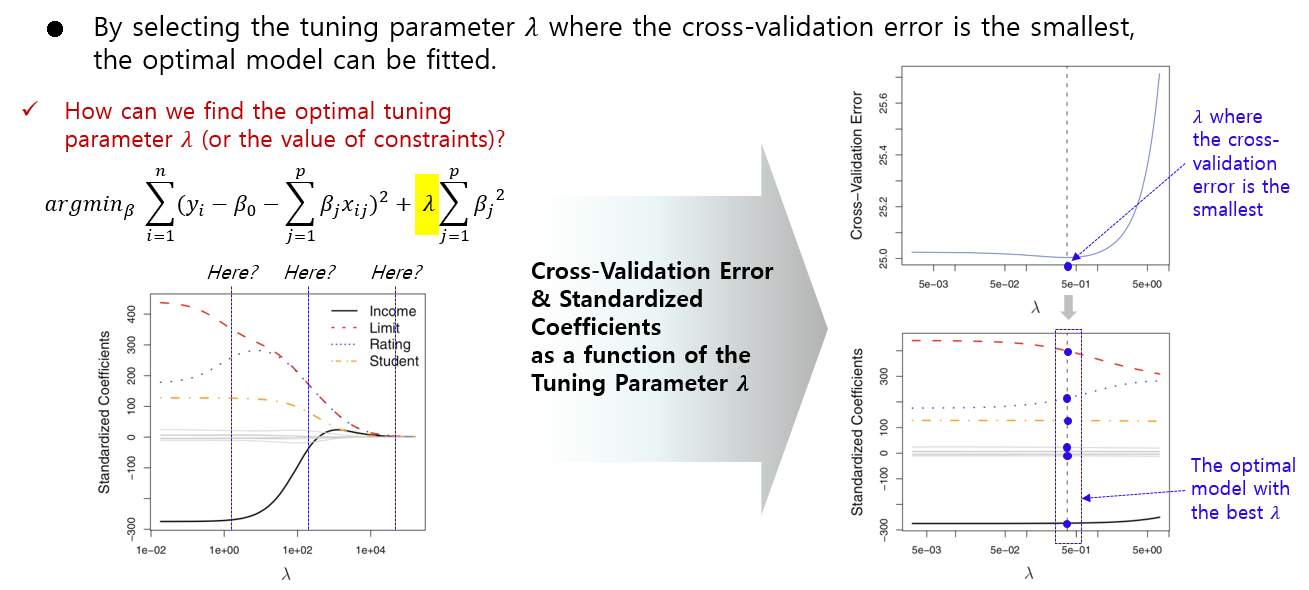
4. Cross-validation을 이용한 Tuning Parameter Gamma 최적값 찾기
5. Python을 이용한 LASSO 예제 (w/ Hyper-parameter tuning)
1. 기계 학습에서의 Regularization 은 무엇이고 왜 사용하는가?
Regularization 은 기계 학습에서 과적합(Overfitting)을 방지하기 위한 기술 중 하나로, 모델이 훈련 데이터에 지나치게 적합화되는 것을 막기 위해 사용됩니다. 과적합은 모델이 훈련 데이터의 잡음과 이상치까지 학습하여 새로운, 보지 못한 데이터에 대해 일반화하기 어렵게 만드는 현상입니다. 이러한 상황에서 모델은 훈련 데이터를 외우기보다는 기본적인 패턴을 학습하는 것이 중요합니다.
Regularization은 모델이 학습하는 파라미터의 계수 (coefficient estimages) 값에 제약을 가해서 '0'으로 가깝게 축소를 하거나 또는 정확하게 '0'으로 축소를 시킴으로써 계수 추정치의 분산을 줄이고(reducing the variance of the coefficients estimates) 모델의 복잡성을 제어하여 과적합을 피하게 해주는 효과가 있습니다. (대신에 Bias 는 늘어나는 비용을 지불하게 됩니다. 세상에 공짜가 없어요..>.<;).
Regularization = Loss Function + Penalty
선형회귀모형을 예로 들어서 설명하면, Regularization 은 Penalty 항을 무엇을 사용하느냐에 따라
(1) L1 Regularization(LASSO), (2) L2 Regularization(Ridge), (3) ElasticNet 으로 구분할 수 있습니다.
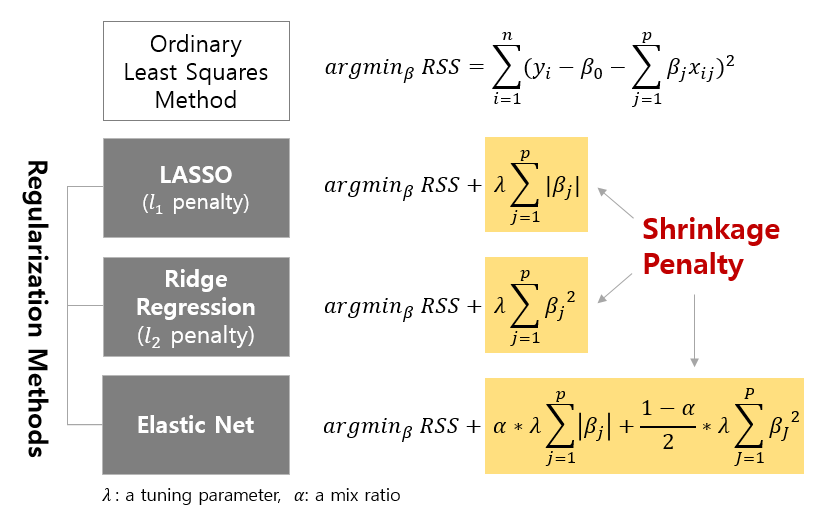
Tuning parameter Lambda(λ)는 모델에 적용되는 규제의 전반적인 강도를 제어하는 규제 강도 하이퍼파라미터입니다. 람다(λ) 값이 높아지면 더 강한 패널티가 적용되어 더 희소한 모델과 작은 계수 값이 생성됩니다.
Elastic Net에 있는 Mix ratio 알파(α) (책에 따라 Alpha 대신 Gamma( γ )가 혼용되서 사용됨. Scikit-Learn 모듈에는 알파(α) 로 표기) 는 Elastic Net에서 L1 및 L2 패널티 항의 조합을 결정하는 하이퍼파라미터입니다.
- 알파(α)가 0이면 Elastic Net은 Ridge 회귀 (L2 Regularization)와 동일합니다.
- 알파(α)가 1이면 Elastic Net은 Lasso 회귀 (L1 Regularization)와 동일합니다.
- 알파(α)가 0과 1 사이의 값을 가지면 Elastic Net은 L1 및 L2 Regularization을 결합합니다.
요약하면, Elastic Net은 알파(α) 매개변수를 통해 L1 및 L2 Regularization을 조합하고, Regularization의 전반적인 강도는 람다(λ) 매개변수로 제어됩니다.
2. L1 Regularization (LASSO Regression*)
LASSO 회귀 또는 L1 Regularization은 모델의 목적 함수에 Penalty 항을 통합하는 선형 회귀 방법입니다. LASSO 회귀의 목표는 과적합을 방지하고 모델이 사용하는 특징의 일부만 사용하도록 유도하여 실질적으로 변수 선택(Feature Selection)을 수행하는 것입니다.
표준 선형 회귀 목적 함수는 예측 값과 실제 값 간의 제곱 차이의 합을 최소화하는 것입니다. 그러나 LASSO 회귀에서는 이 목적 함수에 회귀계수의 절대값의 합이 페널티 항으로 추가되어 수정된 목적 함수는 다음과 같습니다. 즉, 기존의 RSS(Residual Sum of Squares) 에 L1 penalty term(회귀계수의 절대값의 합) 이 더해진 손실함수를 최소로 하는 회계계수 Beta 추정치를 찾는 것입니다. 람다(λ)는 규제하는 강도를 조절하는 매개변수입니다.

이 목적함수 식을 선형계획법(Linear Planning) 의 제약조건(constraints, subject to)을 가지는 식으로 변경해서 다시 써보면 아래의 왼쪽 수식과 같습니다.

* Robert Tibshirani, “Regression Shrinkage and Selection via the Lasso” (1996), Journal of the Royal Statistical Society
페널티 항은 모델에서 일부 특징 가중치를 정확히 0로 축소(shrinks to exactly zero)함으로써 모델 내에서 희소성(sparsity)을 촉진합니다. 이는 모델이 가장 관련성이 높은 특징의 하위 집합을 선택하도록 하고 다른 특징(feature selection)을 무시하도록 만듭니다.
위의 오른쪽 그림은 튜닝 파라미터 람다를 점점 크게 했을 때 변수별 회귀계수의 값이 어느 순간 정확히 0으로 축소되는 것을 알 수 있습니다.
LASSO 회귀는 특히 많은 특징이 존재하며 일부가 무관하거나 중복될 수 있는 상황에서 유용합니다. 희소성을 촉진함으로써 LASSO는 특징 선택(Feature Selection)을 도와주며 해석 가능하고 잠재적으로 간단한 모델을 만듭니다.
요약하면, LASSO 회귀는 L1 Regularization을 사용한 선형 회귀 기술로, 과적합을 방지하고 모델의 특징 가중치에 희소성을 촉진하여 더 나은 일반화 성능을 달성합니다.
3. L2 Regularization (Ridge Regression)
Ridge 회귀 또는 L2 Regularization은 모델의 목적 함수에 패널티 항을 통합하는 선형 회귀 기술입니다. Ridge 회귀의 주요 목적은 선형 회귀 비용 함수의 제곱 계수 합에 페널티 항을 추가함으로써 과적합(Overfitting)을 방지하는 것입니다.
표준 선형 회귀 목적 함수는 예측 값과 실제 값 간의 제곱 차이의 합을 최소화하는 것입니다. 릿지 회귀에서는 이 목적 함수에 페널티 항이 추가되어 수정된 목적은 다음과 같습니다.
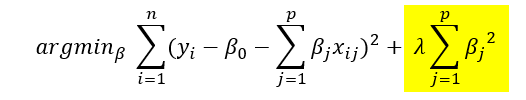
위의 목적함수식을 선형계획법의 제약조건을 가지는 식으로 변경하면 아래와 같습니다.
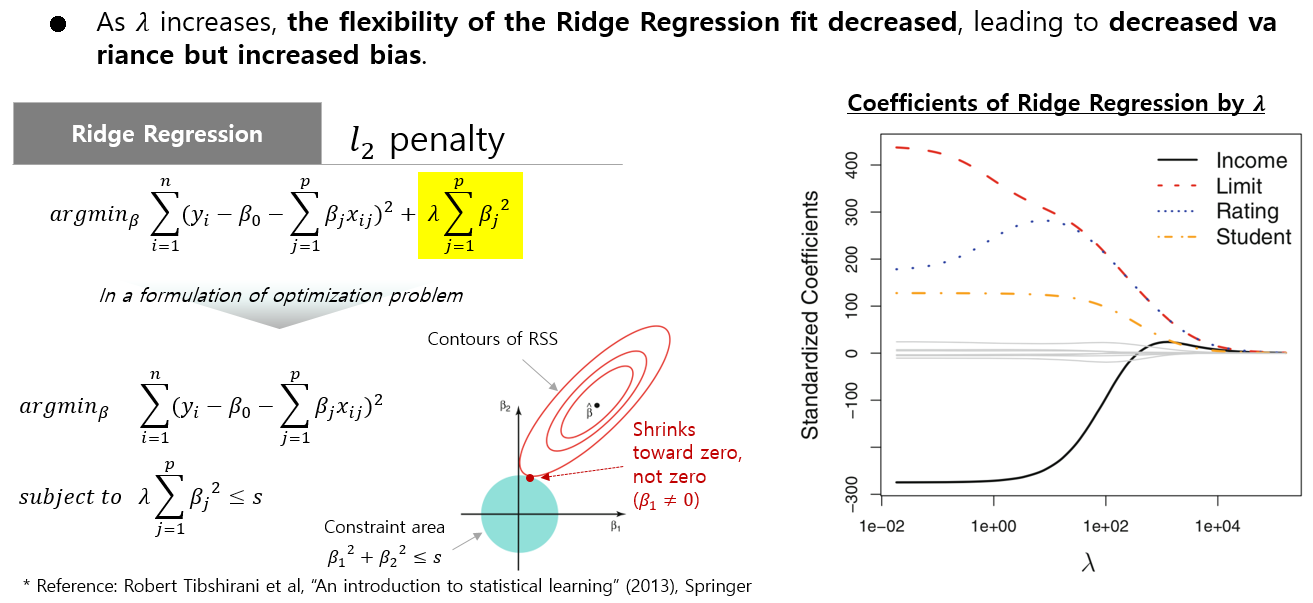
페널티 항은 모델에게 계수를 제로에 가깝게 축소하도록 유도하지만 거의 0으로 만들지는 않습니다.(not exactly zero). 이는 모델이 너무 복잡해지고 훈련 데이터에 과적합되는 것을 방지하는 데 도움이 됩니다.
릿지 회귀는 특징 간에 다중공선성이 있는 경우에 특히 유용하며, 이는 일부 특징이 높은 상관관계를 가질 때 발생합니다. 이러한 경우 릿지 회귀의 정규화 항은 계수를 안정화하고 큰 가중치 값을 피하는 데 도움이 됩니다.
요약하면, 릿지 회귀는 L2 Regularization을 사용한 선형 회귀 기술로, 제곱 계수의 합에 페널티 항을 추가함으로써 오버피팅을 방지합니다. 이는 훈련 데이터를 잘 맞추는 동시에 모델을 간단하게 유지하고 더 일반적으로 만들기 위한 균형을 제공합니다.
4. Cross-validation을 이용한 Tuning Parameter Lambda, Mix ratio Gamma 최적값 찾기
Regularization의 강도를 조절하는 튜닝 파라미터 람다(λ)와 Elastic Net 에서 L1, L2 Regularization의 비율을 조절하는 알파(α)는 데이터로 부터 학습될 수 없고 분석가가 지정해줘야 하는 하이퍼-파라미터(Hyper-parameter) 입니다.
분석 툴에서 제공하는 기본 값을 람다(λ), 알파(α)의 값으로 해도 되지만, 최적의 람다(λ)와 알파(α)를 찾기 위해서는 Cross-validation 기법을 사용합니다.
교차 검증(Cross-validation)은 예측 모델의 성능을 평가하고 과적합의 위험을 줄이기 위한 통계적 기술로, 데이터 집합을 여러 하위 집합으로 나누고 모델을 이러한 하위 집합 중 일부로 훈련하고 나머지로 성능을 평가하는 과정을 포함합니다. 주요 아이디어는 훈련 및 테스트에 서로 다른 하위 집합을 사용하여 모델의 성능에 대한 더 견고한 추정을 얻는 것입니다.
가장 일반적인 교차 검증 형식은 k-겹 교차 검증(k-fold cross-validation)으로, 데이터 집합을 k개의 동일한 크기의 폴드로 나눕니다. 모델은 k번 훈련되며 각 훈련에서 k-1개의 폴드를 사용하고 나머지 폴드를 테스트에 사용합니다. 이 프로세스는 k번 반복되며 각 반복에서 다른 폴드가 테스트에 사용됩니다. 성능 메트릭(예: 정확도, 평균 제곱 오차)은 k번의 반복을 통해 평균화되어 모델의 성능을 더 신뢰할 수 있는 추정으로 제공됩니다.
교차 검증은 특히 하이퍼파라미터 튜닝에 유용하며, 이는 머신 러닝 모델에 대한 최적의 하이퍼파라미터 세트를 찾는 목표입니다. 단일 훈련-테스트 분할을 의존하는 대신 교차 검증을 사용하면 모델의 성능을 여러 훈련-테스트 분할에서 평가할 수 있습니다. 이는 데이터가 특정 랜덤 분할에 따라 평가되는 것에 대한 편향을 줄이는 데 도움이 됩니다.
다음은 하이퍼파라미터 튜닝을 위해 교차 검증을 사용하는 단계별 프로세스입니다:
(1) 데이터 분할: 데이터 집합을 훈련 및 테스트 세트로 나눕니다. 훈련 세트는 k-겹으로 나뉩니다.
(2) 하이퍼파라미터 선택: 튜닝하려는 하이퍼파라미터를 지정합니다.
(3) 하이퍼파라미터 그리드 생성: 튜닝할 하이퍼파라미터 값의 그리드를 정의합니다. 이는 주로 그리드 서치(grid search) 또는 랜덤 서치(random search) 기법을 사용하여 수행됩니다.
(4) 교차 검증 수행: 각 하이퍼파라미터 조합에 대해 k-겹 교차 검증을 수행합니다. k-1개의 폴드를 사용하여 모델을 훈련하고 나머지 폴드를 테스트합니다. 이러한 프로세스를 k번 반복하여 각 반복에서 다른 폴드를 테스트로 사용합니다.
(5) 최적의 하이퍼파라미터 선택: k 폴드를 통해 얻은 성능 중 가장 좋은 평균 성능을 가진 하이퍼파라미터 세트를 선택합니다.
(6) 최종 모델 훈련: 선택된 하이퍼파라미터를 사용하여 전체 훈련 세트에서 최종 모델을 훈련합니다.
교차 검증을 사용하여 하이퍼파라미터 튜닝을 수행하면 데이터의 여러 하위 집합에서 어떤 하이퍼파라미터가 잘 작동하는지에 대한 보다 신뢰할 수 있는 평가를 얻을 수 있습니다.
5. Python sklearn을 이용한 LASSO 예제 (w/ Hyperparameter tuning)
Python의 Scikit-learn 모듈에 있는 sklearn.linear_model.Lasso 메소드를 사용해서 LASSO 회귀를 적합해보겠습니다. 이때 GridSearchCV 로 규제의 강도를 조절하는 튜닝 파라미터 alpha (위에서는 람다로 표기, 좀 헷갈릴 수 있겠네요..) 의 최적의 값을 찾아보도록 하겠습니다.
# Import necessary libraries
import numpy as np
import matplotlib.pyplot as plt
from sklearn.linear_model import Lasso
from sklearn.model_selection import train_test_split, GridSearchCV
from sklearn.metrics import mean_squared_error
# Generate some example data
np.random.seed(42)
x1 = 2 * np.random.rand(100, 1) # strong relation with y
x2 = 2 * np.random.rand(100, 1) # no relation with y
X = np.column_stack([x1, x2])
y = 4 + 3 * x1 + 0.1 * np.random.randn(100, 1)
# Split the data into training and testing sets
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.2, random_state=42)
# Perform Lasso regression with grid search for alpha
lasso = Lasso()
param_grid = {'alpha': [0.01, 0.1, 0.5, 1.0, 2.0]} # Values to try for alpha
grid_search = GridSearchCV(lasso, param_grid, cv=5, scoring='neg_mean_squared_error')
grid_search.fit(X_train, y_train)
# Get the best hyperparameters
best_alpha = grid_search.best_params_['alpha']
# Train the model with the best hyperparameters
lasso_reg = Lasso(alpha=best_alpha)
lasso_reg.fit(X_train, y_train)
# Read out attributes
coeffs = lasso_reg.coef_ # coefficients
intercept = lasso_reg.intercept_ # intercept
# Make predictions on the test set
y_pred = lasso_reg.predict(X_test)
# Evaluate the model
mse = mean_squared_error(y_test, y_pred)
print(f'Best alpha: {best_alpha}')
print('Coefficients:', coeffs)
print('Intercept:', intercept)
print(f'Mean Squared Error: {mse}')
# Best alpha: 0.01
# Coefficients: [2.95614483 0.00726315]
# Intercept: [4.04106647]
# Mean Squared Error: 0.0066267863966506645
Cross-validation 으로 찾은 최적의 best alpha 는 0.01 이네요.
Alpha = 0.01 일 때 목표변수 y와 강한 상관성이 있는 변수 x1의 회귀계수는 2.956이고, 목표변수 y와는 아무런 상관이 없는 변수 x2 의 회귀계수는 0.0072 로서 거의 0 으로 축소되었습니다.
이번 포스팅이 많은 도움이 되었기를 바랍니다.
행복한 데이터 과학자 되세요! :-)