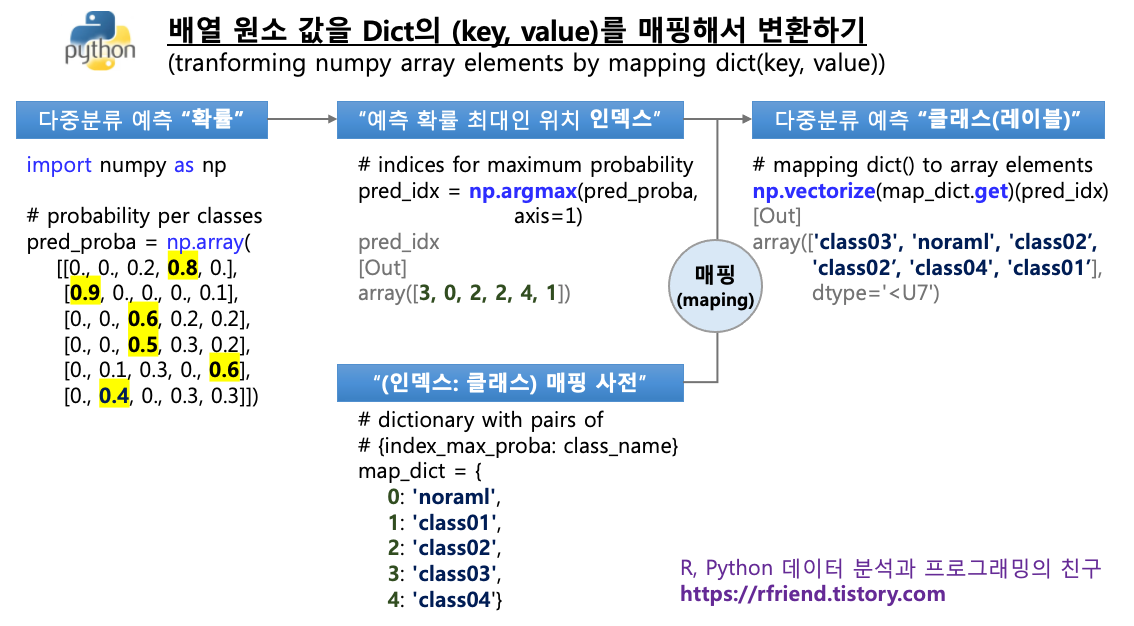
[Python] DataFrame에서 여러개의 변수에 대해 일원분산분석 검정하기 (ANOVA test for multiple numeric variables in pandas DataFrame)
Python 분석과 프로그래밍/Python 통계분석 2021. 5. 8. 19:22지난번 포스팅에서는 샘플 크기가 다른 2개 이상의 집단에 대해 평균의 차이가 존재하는지를 검정하는 일원분산분석(one-way ANOVA)에 대해 scipy 모듈의 scipy.stats.f_oneway() 메소드를 사용해서 분석하는 방법(rfriend.tistory.com/638)을 소개하였습니다.
이번 포스팅에서는 2개 이상의 집단에 대해 pandas DataFrame에 들어있는 여러 개의 숫자형 변수(one-way ANOVA for multiple numeric variables in pandas DataFrame) 별로 일원분산분석 검정(one-way ANOVA test)을 하는 방법을 소개하겠습니다.
숫자형 변수와 집단 변수의 모든 가능한 조합을 MultiIndex 로 만들어서 statsmodels.api 모듈의 stats.anova_lm() 메소드의 모델에 for loop 순환문으로 변수를 바꾸어 가면서 ANOVA 검정을 하도록 작성하였습니다.
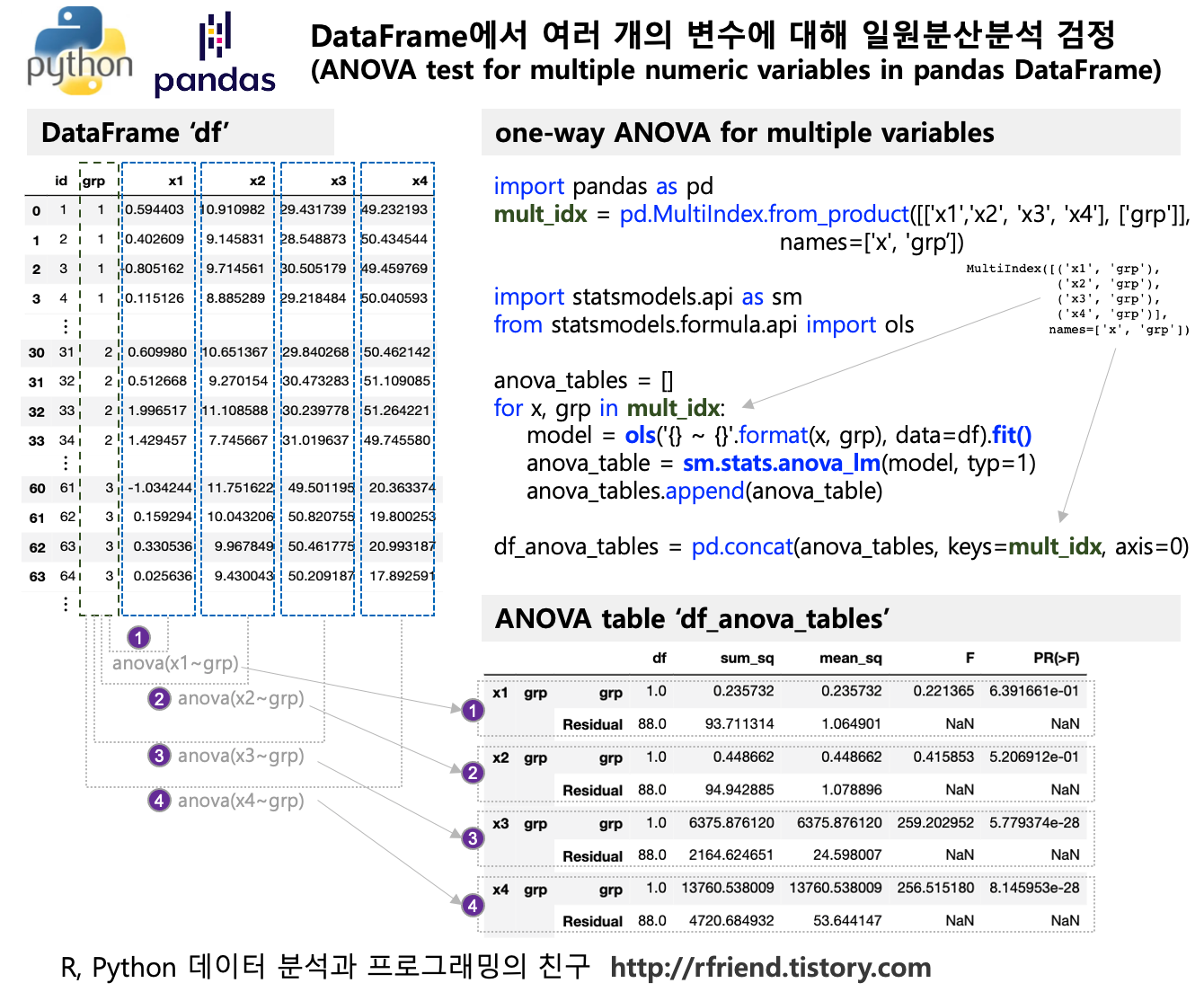
먼저, 3개의 집단('grp 1', 'grp 2', 'grp 3')을 별로 'x1', 'x2', 'x3, 'x4' 의 4개의 숫자형 변수를 각각 30개씩 가지는 가상의 pandas DataFrame을 만들어보겠습니다. 이때 숫자형 변수는 모두 정규분포로 부터 난수를 발생시켜 생성하였으며, 'x3'와 'x4'에 대해서는 집단3 ('grp 3') 의 평균이 다른 2개 집단의 평균과는 다른 정규분포로 부터 난수를 발생시켜 생성하였습니다.
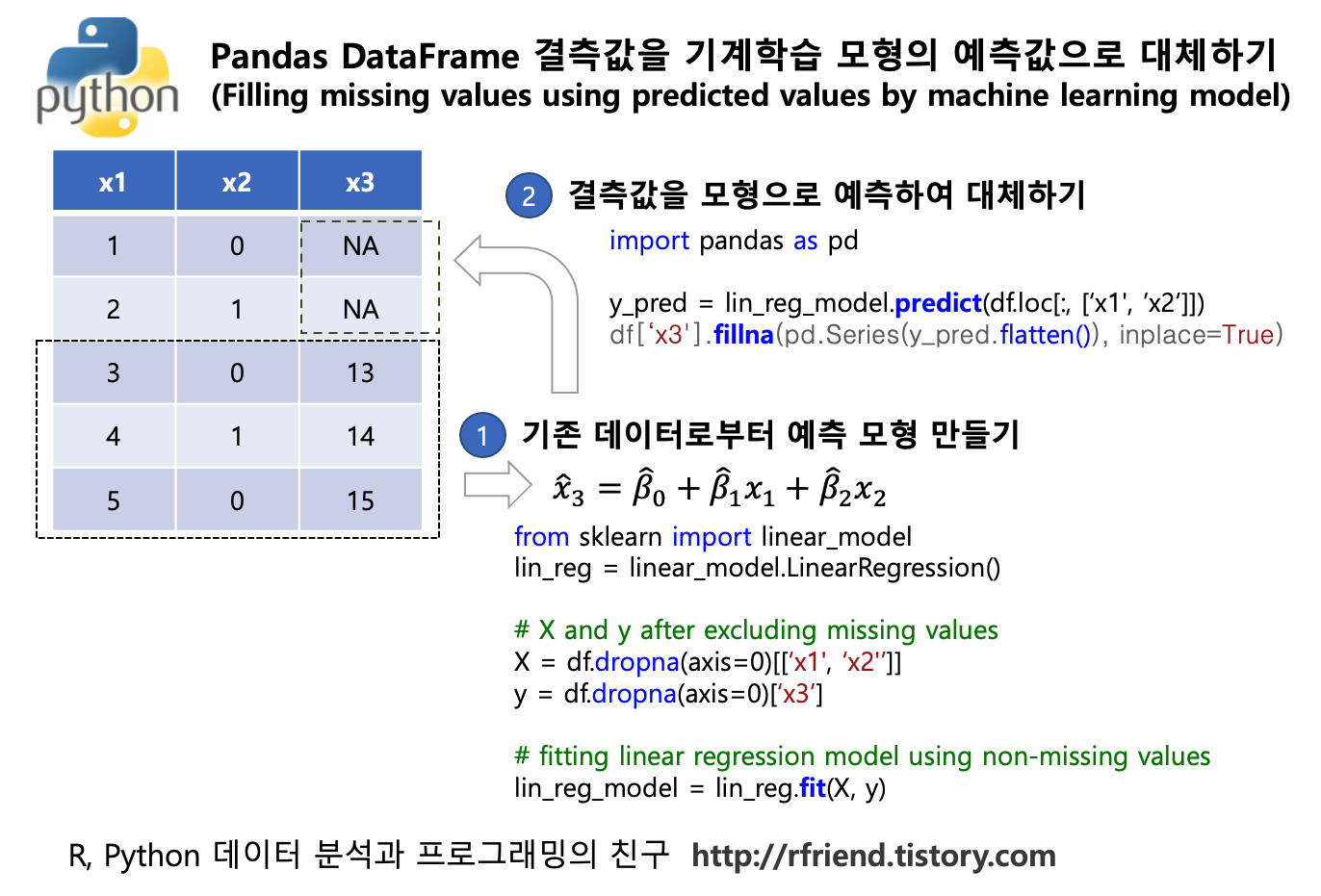
아래의 가상 데이터셋은 결측값이 없이 만들었습니다만, 실제 기업에서 쓰는 데이터셋에는 혹시 결측값이 존재할 수도 있으므로 결측값을 없애거나 또는 결측값을 그룹 별 평균으로 대체한 후에 one-way ANOVA 를 실행하기 바랍니다.
## Creating sample dataset
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
import seaborn as sns
# generate 90 IDs
id = np.arange(90) + 1
# Create 3 groups with 30 observations in each group.
from itertools import chain, repeat
grp = list(chain.from_iterable((repeat(number, 30) for number in [1, 2, 3])))
# generate random numbers per each groups from normal distribution
np.random.seed(1004)
# for 'x1' from group 1, 2 and 3
x1_g1 = np.random.normal(0, 1, 30)
x1_g2 = np.random.normal(0, 1, 30)
x1_g3 = np.random.normal(0, 1, 30)
# for 'x2' from group 1, 2 and 3
x2_g1 = np.random.normal(10, 1, 30)
x2_g2 = np.random.normal(10, 1, 30)
x2_g3 = np.random.normal(10, 1, 30)
# for 'x3' from group 1, 2 and 3
x3_g1 = np.random.normal(30, 1, 30)
x3_g2 = np.random.normal(30, 1, 30)
x3_g3 = np.random.normal(50, 1, 30) # different mean
x4_g1 = np.random.normal(50, 1, 30)
x4_g2 = np.random.normal(50, 1, 30)
x4_g3 = np.random.normal(20, 1, 30) # different mean
# make a DataFrame with all together
df = pd.DataFrame({'id': id,
'grp': grp,
'x1': np.concatenate([x1_g1, x1_g2, x1_g3]),
'x2': np.concatenate([x2_g1, x2_g2, x2_g3]),
'x3': np.concatenate([x3_g1, x3_g2, x3_g3]),
'x4': np.concatenate([x4_g1, x4_g2, x4_g3])})
df.head()
[Out]
id grp x1 x2 x3 x4
0 1 1 0.594403 10.910982 29.431739 49.232193
1 2 1 0.402609 9.145831 28.548873 50.434544
2 3 1 -0.805162 9.714561 30.505179 49.459769
3 4 1 0.115126 8.885289 29.218484 50.040593
4 5 1 -0.753065 10.230208 30.072990 49.601211
df[df['grp'] == 3].head()
[Out]
id grp x1 x2 x3 x4
60 61 3 -1.034244 11.751622 49.501195 20.363374
61 62 3 0.159294 10.043206 50.820755 19.800253
62 63 3 0.330536 9.967849 50.461775 20.993187
63 64 3 0.025636 9.430043 50.209187 17.892591
64 65 3 -0.092139 12.543271 51.795920 18.883919
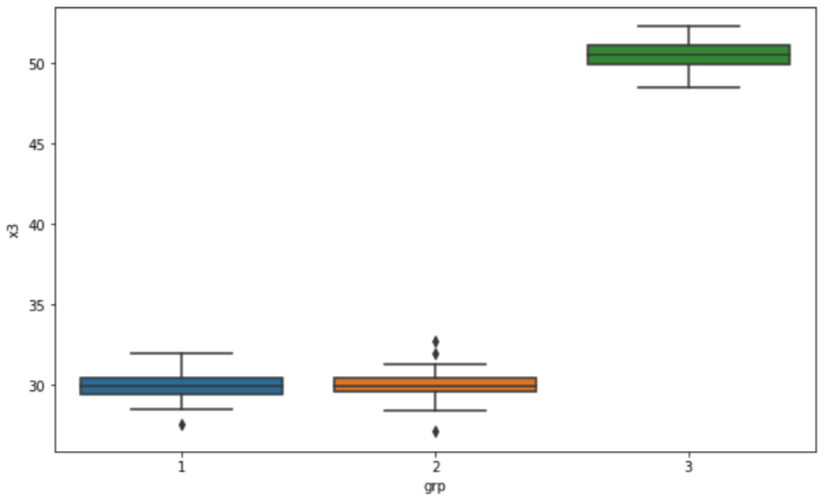
가령, 'x3' 변수에 대해 집단별로 상자 그래프 (Box plot for 'x3' by groups) 를 그려보면, 아래와 같이 집단1과 집단2는 유사한 반면에 집단3은 평균이 차이가 많이 나게 가상의 샘플 데이터가 생성되었음을 알 수 있습니다.
## Boxplot for 'x3' by 'grp'
plt.rcParams['figure.figsize'] = [10, 6]
sns.boxplot(x='grp', y='x3', data=df)
plt.show()
여러개의 변수에 대해 일원분산분석을 하기 전에, 먼저 이해를 돕기 위해 Python의 statsmodels.api 모듈의 stats.anova_lm() 메소드를 사용해서 'x1' 변수에 대해 집단(집단 1/2/3)별로 평균이 같은지 일원분산분석으로 검정을 해보겠습니다.
- 귀무가설(H0) : 집단1의 x1 평균 = 집단2의 x1 평균 = 집단3의 x1 평균
- 대립가설(H1) : 적어도 1개 이상의 집단의 x1 평균이 다른 집단의 평균과 다르다. (Not H0)
# ANOVA for x1 and grp
import statsmodels.api as sm
from statsmodels.formula.api import ols
model = ols('x1 ~ grp', data=df).fit()
sm.stats.anova_lm(model, typ=1)
[Out]
df sum_sq mean_sq F PR(>F)
grp 1.0 0.235732 0.235732 0.221365 0.639166
Residual 88.0 93.711314 1.064901 NaN NaN
일원분산분석 결과 F 통계량이 0.221365, p-value가 0.639 로서 유의수준 5% 하에서 귀무가설을 채택합니다. 즉, 3개 집단 간 x1의 평균의 차이는 없다고 판단할 수 있습니다. (정규분포 X ~ N(0, 1) 를 따르는 모집단으로 부터 무작위로 3개 집단의 샘플을 추출했으므로 차이가 없게 나오는게 맞겠습니다.)
한개의 변수에 대한 일원분산분석하는 방법을 알아보았으니, 다음으로는 3개 집단별로 여러개의 연속형 변수인 'x1', 'x2', 'x3', 'x4' 에 대해서 for loop 순환문으로 돌아가면서 일원분산분석을 하고, 그 결과를 하나의 DataFrame에 모아보도록 하겠습니다.
(1) 먼저, 일원분산분석을 하려는 모든 숫자형 변수와 집단 변수에 대한 가능한 조합의 MultiIndex 를 생성해줍니다.
# make a multiindex for possible combinations of Xs and Group
num_col = ['x1','x2', 'x3', 'x4']
cat_col = ['grp']
mult_idx = pd.MultiIndex.from_product([num_col, cat_col],
names=['x', 'grp'])
print(mult_idx)
[Out]
MultiIndex([('x1', 'grp'),
('x2', 'grp'),
('x3', 'grp'),
('x4', 'grp')],
names=['x', 'grp'])
(2) for loop 순환문(for x, grp in mult_idx:)으로 model = ols('{} ~ {}'.format(x, grp) 의 선형모델의 y, x 부분의 변수 이름을 바꾸어가면서 sm.stats.anova_lm(model, typ=1) 로 일원분산분석을 수행합니다. 이렇게 해서 나온 일원분산분석 결과 테이블을 anova_tables.append(anova_table) 로 순차적으로 append 해나가면 됩니다.
# ANOVA test for multiple combinations of X and Group
import statsmodels.api as sm
from statsmodels.formula.api import ols
anova_tables = []
for x, grp in mult_idx:
model = ols('{} ~ {}'.format(x, grp), data=df).fit()
anova_table = sm.stats.anova_lm(model, typ=1)
anova_tables.append(anova_table)
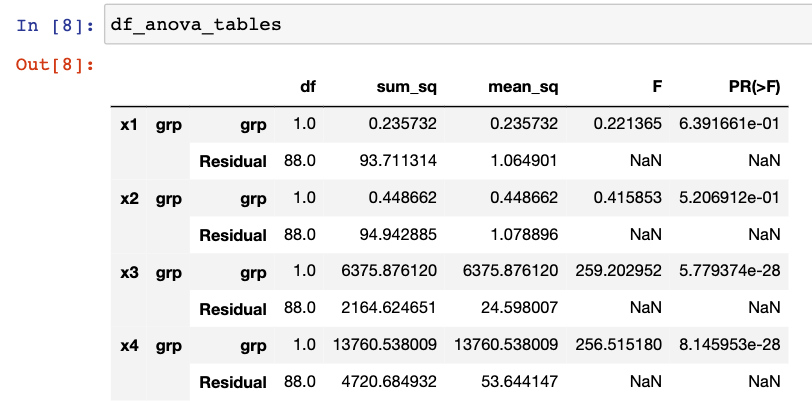
df_anova_tables = pd.concat(anova_tables, keys=mult_idx, axis=0)
df_anova_tables
[Out]
df sum_sq mean_sq F PR(>F)
x1 grp grp 1.0 0.235732 0.235732 0.221365 6.391661e-01
Residual 88.0 93.711314 1.064901 NaN NaN
x2 grp grp 1.0 0.448662 0.448662 0.415853 5.206912e-01
Residual 88.0 94.942885 1.078896 NaN NaN
x3 grp grp 1.0 6375.876120 6375.876120 259.202952 5.779374e-28
Residual 88.0 2164.624651 24.598007 NaN NaN
x4 grp grp 1.0 13760.538009 13760.538009 256.515180 8.145953e-28
Residual 88.0 4720.684932 53.644147 NaN NaN
만약 특정 변수에 대한 일원분산분석 결과만을 조회하고 싶다면, 아래처럼 DataFrame의 MultiIndex 에 대해 인덱싱을 해오면 됩니다. 가령, 'x3' 에 대한 집단별 평균 차이 여부를 검정한 결과는 아래처럼 인덱싱해오면 됩니다.
## Getting values of 'x3' from ANOVA tables
df_anova_tables.loc[('x3', 'grp', 'grp')]
[Out]
df 1.000000e+00
sum_sq 6.375876e+03
mean_sq 6.375876e+03
F 2.592030e+02
PR(>F) 5.779374e-28
Name: (x3, grp, grp), dtype: float64
F 통계량과 p-value 에 대해서 조회하고 싶으면 위의 결과에서 DataFrame 의 칼럼 이름으로 선택해오면 됩니다.
# F-statistic
df_anova_tables.loc[('x3', 'grp', 'grp')]['F']
[Out]
259.2029515179077
# P-value
df_anova_tables.loc[('x3', 'grp', 'grp')]['PR(>F)']
[Out]
5.7793742588216585e-28
MultiIndex 를 인덱싱해오는게 좀 불편할 수 도 있는데요, 이럴 경우 df_anova_tables.reset_index() 로 MultiIndex 를 칼럼으로 변환해서 사용할 수도 있습니다.
# resetting index to columns
df_anova_tables_2 = df_anova_tables.reset_index().dropna()
df_anova_tables_2
[Out]
level_0 level_1 level_2 df sum_sq mean_sq F PR(>F)
0 x1 grp grp 1.0 0.235732 0.235732 0.221365 6.391661e-01
2 x2 grp grp 1.0 0.448662 0.448662 0.415853 5.206912e-01
4 x3 grp grp 1.0 6375.876120 6375.876120 259.202952 5.779374e-28
6 x4 grp grp 1.0 13760.538009 13760.538009 256.515180 8.145953e-28
Greenplum DB에서 PL/Python (또는 PL/R)을 사용하여 여러개의 숫자형 변수에 대해 일원분산분석을 분산병렬처리하는 방법(one-way ANOVA in parallel using PL/Python on Greenplum DB)은 rfriend.tistory.com/640 를 참고하세요.
[reference]
* ANOVA test using Python statsmodels
: https://www.statsmodels.org/stable/generated/statsmodels.stats.anova.anova_lm.html
이번 포스팅이 많은 도움이 되었기를 바랍니다.
행복한 데이터 과학자 되세요! :-)